Sommario:
- Passaggio 1: utilizzo di Brain Box
- Passaggio 2: introduzione: analisi delle sezioni trasversali
- Passaggio 3: impostazione di una funzione: LevelCurveTracings.m
- Passaggio 4: sviluppo dell'algoritmo Bound: Trovare i limiti
- Passaggio 5: sviluppo dell'algoritmo Bound: generazione di un array esterno esterno
- Passaggio 6: sviluppo dell'algoritmo associato: lavorare con il punto centrale
- Passaggio 7: sviluppo dell'algoritmo legato: quando un centroide non è centrato
- Passaggio 8: sviluppo dell'algoritmo legato: interferenza dei fori
- Passaggio 9: sviluppo dell'algoritmo Bound: Individuazione dei fori, finalizzazione dei limiti del cervello e dei limiti dei fori
- Passaggio 10: registrazione dei dati: funzione PatientFiles.m
- Passaggio 11: registrazione dei dati nei file
- Passaggio 12: registrazione dei dati: visualizzazione di un grafico del volume del cervello nel tempo
- Passaggio 13: colmare le lacune nelle sottotrame: Subplotclose.m
- Passaggio 14: Il codice principale: cancellazione di tutto e richiesta di input
- Passaggio 15: Il codice principale: elaborazione in batch delle immagini
- Passaggio 16: Il codice principale: Imbottitura
- Passaggio 17: Il codice principale: determinazione dei confini
- Passaggio 18: Il codice principale: determinare la corretta dimensione Z
- Passaggio 19: Il codice principale: determinazione delle coordinate X e Y
- Passaggio 20: Il codice principale: tracciare una struttura tridimensionale, trovare il volume e registrare i dati
- Autore John Day day@howwhatproduce.com.
- Public 2024-01-31 10:21.
- Ultima modifica 2025-01-23 14:50.
L'avanzata verso la frontiera di una vita umana più lunga ha portato alla comparsa di malattie mai viste da civiltà prima della nostra. Tra questi, l'Alzheimer ha colpito circa 5,3 milioni di americani anziani viventi nel 2017, o circa 1 americano anziano su 10 (https://www.alz.org/facts/) e innumerevoli altri con demenza. Per aiutare nella lotta per capire cosa sta affliggendo i nostri anziani, questo codice doterà i futuri ricercatori e gli ansiosi curiosi della capacità di monitorare il volume del cervello nel tempo.
Passaggio 1: utilizzo di Brain Box
Per utilizzare la scatola del cervello, è necessario solo quanto segue:
- Scansioni MRI di un cervello e il nome e il formato di tali file (dovrebbero avere tutti più o meno le stesse dimensioni)
- Lunghezza di una scansione
- Distanza tra ogni strato (scansione MRI)
- Nome del paziente (durante l'inserimento, non includere spazi e si prega di scrivere nome e cognome in maiuscolo, in questo modo: NomeCognome)
E da questo, si ha la capacità di tenere traccia delle tendenze di un individuo nel volume del cervello nel tempo. Pertanto, questo software può tenere traccia delle cifre relative alle tendenze dell'Alzheimer. La lunghezza che abbiamo usato nella prova era di 180 mm per la lunghezza di una scansione e 5 mm per la distanza tra le scansioni MRI, basata su cifre medie.
Tuttavia, l'applicazione della scatola del cervello non deve essere vincolata a questo compito. Se le sezioni trasversali di un determinato solido sono fotografate, come un tumore di per sé, le tendenze nelle variazioni di volume per questi possono anche essere monitorate nel software.
Passaggio 2: introduzione: analisi delle sezioni trasversali
Nelle strutture tridimensionali, i piani bidimensionali di cui sono costituiti sarebbero chiamati sezioni trasversali. Immagina che una pila di fogli costituisca un prisma rettangolare, quindi ogni pezzo di carta sarebbe una sezione trasversale della carta. Nell'immaginare il cervello, applichiamo lo stesso corso di pensiero. La risonanza magnetica (risonanza magnetica) (vedi informazioni sulla risonanza magnetica) cattura le sezioni trasversali del cervello e, utilizzando i confini definiti in ogni "strato" del cervello fornito, possiamo costruire una struttura per modellare e trovare il volume del cervello. Tuttavia, dobbiamo prima costruire una funzione per fornire informazioni su tali confini.
Passaggio 3: impostazione di una funzione: LevelCurveTracings.m
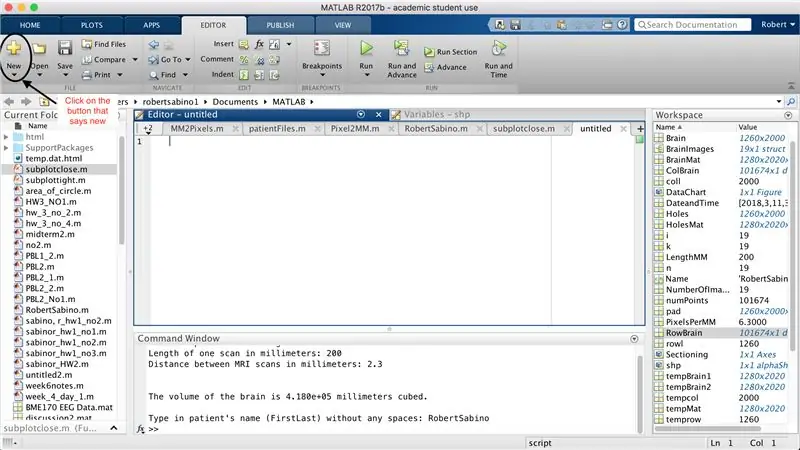
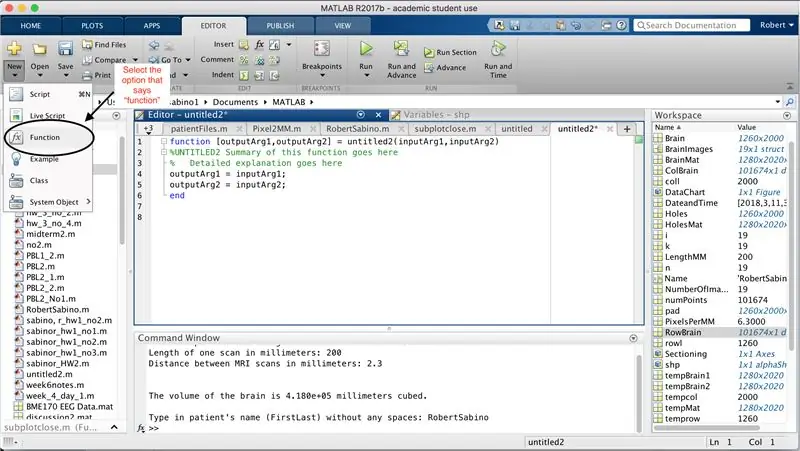
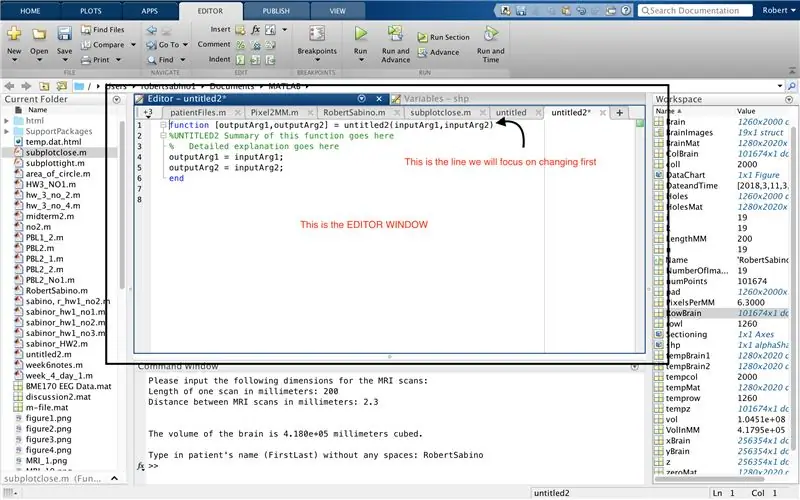

Innanzitutto, assicurati che sul tuo computer sia stato scaricato MATLAB_R2017b (scarica qui) e apri MATLAB. Nell'interfaccia di MATLAB, fare clic sul pulsante nell'angolo in alto a sinistra della finestra che dice "Nuovo" con un segno più giallo in grassetto e selezionare l'opzione "funzione", per aprire nella finestra dell'editor uno spazio simile a quello nel terza foto. Ci concentreremo sulla modifica della prima riga per impostare la funzione. Dove dice "outputArg1", sostituiscilo con "brain", "outputArg2" per dire "holes", "untitled2" per "exp2" e "inputArg1" per "image" ed elimina "inputArg2". Ora hai una funzione da chiamare usando "exp2", prendendo un argomento "image" ed emettendo i confini di "brain" e "holes". La prima riga della funzione dovrebbe assomigliare alla riga rappresentata nella quarta immagine. Elimina tutto il codice sotto questa riga iniziale.
Passaggio 4: sviluppo dell'algoritmo Bound: Trovare i limiti

Digitare il codice come segue sotto la riga. Questa sezione della funzione esegue le seguenti operazioni riga per riga.
- Carica l'immagine "image" nella variabile "mri".
- Trasforma "mri" in un'immagine composta da valori in un intervallo di numeri a uno e zero (noto anche come binarizzazione) in base a un valore soglia impostato. Se il valore in un pixel è uguale o maggiore di 0,1, viene impostato su uno, in caso contrario, il valore in quel pixel viene impostato su zero.
- Le seguenti quattro righe trasformano in zeri 10 colonne e righe ai bordi del livello di scansione MRI, per evitare di leggere valori impropri come creazione di limiti (come appreso sperimentando il codice).
- Nella riga finale, bwboundaries traccia i limiti dell'immagine binarizzata "mri" e la pone uguale a "b", un array con gli elementi i cui indici corrispondono a quelli dei limiti impostati a uno.
Passaggio 5: sviluppo dell'algoritmo Bound: generazione di un array esterno esterno
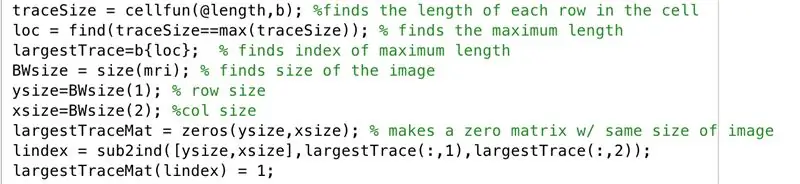
Segui nella finestra dell'editor con il seguente codice nell'immagine. Questa sezione del codice esegue la seguente riga per riga.
- Trova la lunghezza di ciascuna delle righe dell'immagine binarizzata "b" (cellfun applica la lunghezza della funzione a ciascuna riga).
- Impostare "loc" per memorizzare le lunghezze massime.
- Trova l'indice della lunghezza massima, impostato per memorizzare in "largestTrace".
- Trova la dimensione dell'immagine "mri", che ha le stesse dimensioni di "b", e impostala su "BWsize".
- Trova il numero di righe nell'array dell'immagine, impostalo su "ysize".
- Trova il numero di colonne nell'array dell'immagine, impostato su "xsize".
- Genera array "largestTraceMat", una matrice di zeri "ysize" per "xsize".
- Trova l'indice equivalente dai valori in pedice corrispondenti a dove erano i valori più grandi di Trace x e y, memorizza nel vettore "lindex".
- Nella matrice di zeri, "largestTraceMat", trasforma in unità gli elementi negli indici che corrispondono ai valori dell'indice memorizzati come elementi in "lindex".
Pertanto l'array logico "largestTraceMat" ha la regione delimitata più grande della data sezione trasversale della scansione cerebrale tracciata come unità con uno sfondo di zeri
Passaggio 6: sviluppo dell'algoritmo associato: lavorare con il punto centrale

Successivamente, dobbiamo verificare se la sezione trasversale è composta da più di una regione (la più grande). Testando l'allineamento del centroide della regione più grande, possiamo vedere se c'è una regione contigua, che produrrebbe un centroide più centrato, o la possibilità di più regioni.
- Usa "regionProps" per trovare informazioni sui centroidi presenti, impostato uguale all'array della struttura "tempStruct"
- Form array "centroidi" con i dati del campo "centroide" concatenati verticalmente
- Prendi i valori della seconda colonna di "centroidi" (le coordinate della dimensione orizzontale)
- Eseguire un filtro per verificare l'allineamento del baricentro rispetto al centro orizzontale
Passaggio 7: sviluppo dell'algoritmo legato: quando un centroide non è centrato
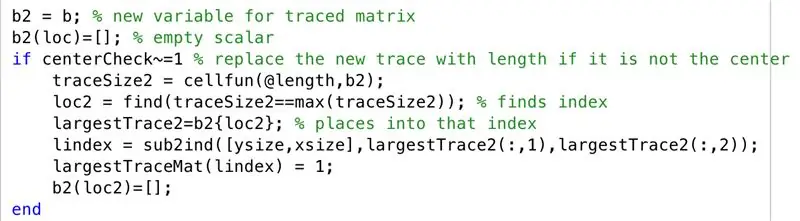
Nello scenario in cui il centroide della regione di traccia più grande non è centrato, seguiamo i passaggi seguenti. Come avevamo osservato nelle scansioni MRI, la tendenza era quella di avere emisferi del cervello raffigurati nella sezione trasversale quando non contigui, quindi ora continuiamo a tracciare la seconda traccia più grande insieme alla traccia più grande in "largestTraceMat"
- Imposta la matrice tracciata su una nuova variabile "b2"
- Inizializza la matrice vuota "b2", con un set indicizzato da "loc"
- Crea un condizionale, per quando un centroide non è centrato (ovvero un layer con più regioni)
- Imposta una nuova dimensione di traccia da trovare per ogni riga (traceSize2)
- Imposta "loc2" per trovare gli indici in cui sono presenti i limiti
- Lascia che le celle specificate da "loc2" in "b2" siano uguali a "largestTrace2"
- Converti i pedici in indici, imposta su "lindex"
- Cambia gli elementi corrispondenti a "lindex" in "largestTraceMat" a 1
- Inizializza la matrice vuota "b2", con un set indicizzato da "loc2"
Passaggio 8: sviluppo dell'algoritmo legato: interferenza dei fori

Nell'affrontare i buchi, i valori memorizzati in "b2" hanno tenuto traccia di strutture diverse dalla traccia più grande e il loro tracciamento su una forma piena di "largestTraceMat" rivelerà dove ci sono buchi nelle regioni del cervello.
- Crea l'array "filledMat", che è un modulo compilato di "largestTraceMat"
- Crea un array "interferenceMat", un array di zeri "ysize" per "xsize"
- Crea array "interferenceloc", per memorizzare i valori da "b2", concatenati verticalmente
- Crea array "lindex" per memorizzare gli indici che corrispondono a "interferenceloc"
- Per gli indici in "interferenceMat" che corrispondono a "lindex", impostare il valore su 1, creando una regione delimitata diversa
Passaggio 9: sviluppo dell'algoritmo Bound: Individuazione dei fori, finalizzazione dei limiti del cervello e dei limiti dei fori

- Imposta l'array "tempMat" uguale a "interferenceMat" più "filledMat", aggiungendo così ogni valore nella matrice l'uno all'altro
- Imposta l'array "holesLoc" uguale agli indici in cui "interferenceMat" e "fillMat" erano entrambi uguali a uno
- Imposta "holesMat" come matrice zero di dimensioni "ysize" x "xsize"
- Imposta gli indici in "holesMat" che sono uguali a "holesLoc" come unità
- Imposta "cervello" su "più grandeTraceMat"
- Imposta "holes" su "holesMat"
Con la scoperta in cui i valori delle matrici aggiunte erano uguali a 2, le posizioni dei fori sono state facilmente fissate e tracciate su una matrice vuota.
Passaggio 10: registrazione dei dati: funzione PatientFiles.m
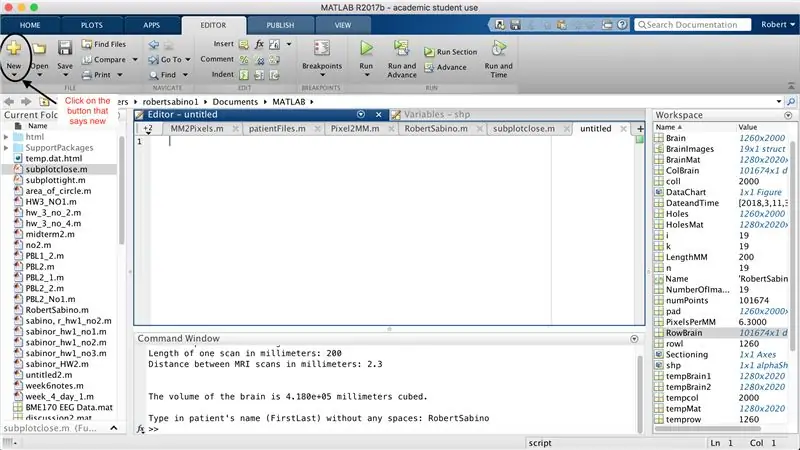
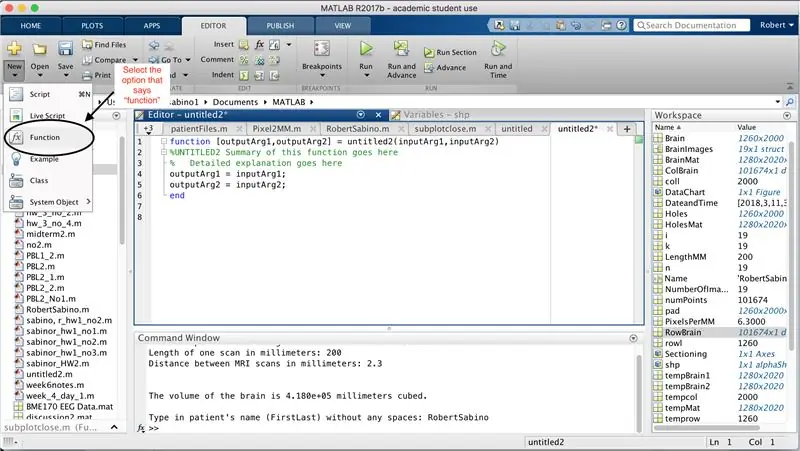
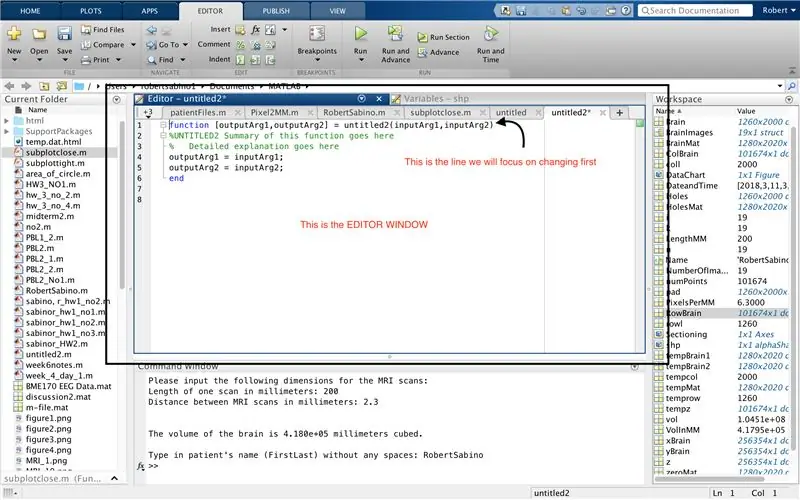

Proprio come l'impostazione dell'ultima funzione, fare clic sul pulsante nell'angolo in alto a sinistra della finestra che dice "Nuovo" con un segno più giallo in grassetto e selezionare l'opzione "funzione", per aprire nella finestra dell'editor uno spazio che assomiglia a quello della terza foto. Nella prima riga, elimina la matrice di output e sostituisci con semplicemente "output", sostituisci "untitled2" con "patientFiles", elimina tutti gli argomenti di input e segui invece la formattazione specificata nella quarta immagine della riga di codice. La prima riga di questa funzione dovrebbe corrispondere alla formattazione dell'immagine.
Passaggio 11: registrazione dei dati nei file
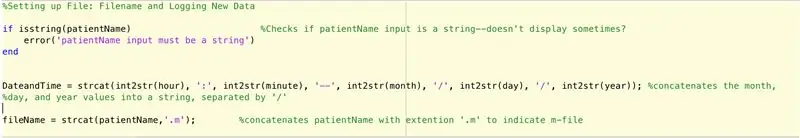

Per impostare un file per registrare i dati trovati dalla funzione principale (ancora da descrivere), dobbiamo seguire questi passaggi (come prescritto dal codice riga per riga).
- Controllare se l'input per il nomepaziente è una stringa.
- Se non è una stringa, visualizzare che l'input di PatientName dovrebbe essere una stringa.
- Termina l'istruzione if (previeni l'errore).
- Imposta un'istruzione stringa "DateandTime" che dia il seguente formato: ora:minuti--mese/giorno/anno.
- Imposta la variabile fileName su quanto segue: nomePaziente.m.
Passiamo ora alla sezione successiva della funzione: esiste già un file con questo nome?
1) Supponiamo che il file con questo nome esista già:
- Esegui il file per ottenere i valori del passato in coda
- Aggiungi i dati "DateandTime" dell'iterazione corrente come una nuova cella nell'array di celle di x valori (fine indice+1)
- Aggiungi il valore "brainVolume" corrente come nuova cella nell'array di celle di valori y (fine indice+1)
- Salva le variabili correnti caricate nel file.
2) Supponiamo che il file con questo nome non esista:
- Crea un nuovo file con il nome memorizzato nella variabile "PazienteName"
- Aggiungi i dati "DateandTime" correnti come cella nell'array di celle vuote di x valori
- Aggiungi i dati "brainVolume" correnti come cella nell'array di celle vuote di valori y
- Salva le variabili correnti caricate nel file.
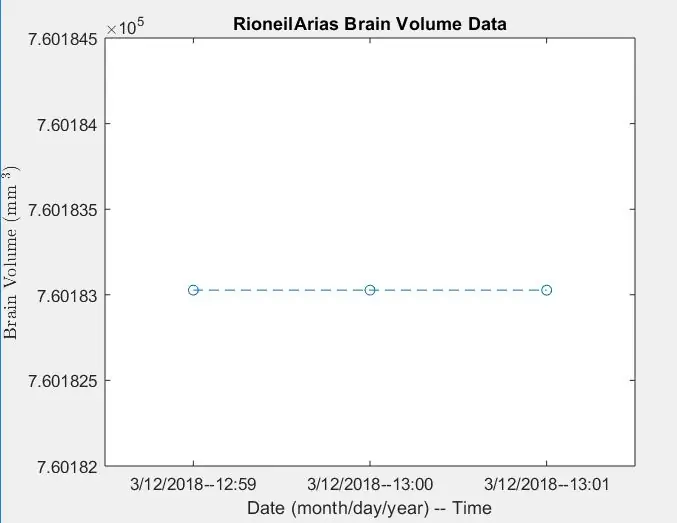
Passaggio 12: registrazione dei dati: visualizzazione di un grafico del volume del cervello nel tempo

- Converti l'array di valori x (xVals) in un array categorico (xValsCategorical), per consentire il tracciamento
- Finestra di generazione della figura 5
- Traccia i punti designati da "xValsCategorical" e "yVals" (contenenti il volume del cervello), usando cerchi vuoti per indicare i punti e da collegare da linee tratteggiate
- Intitola la trama come: PatientName Brain Volume Data
- Etichetta l'asse x come mostrato nell'immagine
- Etichetta l'asse y come mostrato nell'immagine
- Sia la figura 5 uguale all'output
Da questo, la funzione PatientName che viene chiamata produrrà un file con dati modificati che tengono traccia del volume del cervello nel tempo e un grafico che mostra le tendenze.
Passaggio 13: colmare le lacune nelle sottotrame: Subplotclose.m

La funzione, adattata dal codice da https://www.briandalessandro.com, funziona per colmare le lacune tra le figure della sottotrama del codice principale, quando vengono create le figure che mostrano le immagini MRI e gli strati del cervello. La funzione di sottotrama utilizzata all'interno di subplotclose.m regola la posizione delle sottotrame date per adattarsi perfettamente l'una all'altra nell'aspetto della dimensione più lunga. Ad esempio, se il codice intende una matrice 7 x 3, le righe si adatteranno perfettamente poiché la dimensione della riga è più lunga. Se il codice intende una matrice 3 x 7, le colonne si adatteranno perfettamente, con spazi nelle righe, come mostrato nelle figure del nostro codice principale.
Passaggio 14: Il codice principale: cancellazione di tutto e richiesta di input

Per avviare il codice principale, fai clic sullo stesso pulsante che dice "Nuovo" nell'angolo in alto a sinistra della finestra e seleziona "Script" invece di "Funzione" dalle sezioni precedenti. Digita il codice come mostrato nell'immagine all'interno della finestra dell'editor. Le righe di codice svolgono le seguenti attività in ordine:
- Chiudi tutti i file aperti tranne 0, 1 e 2.
- Chiudi tutte le finestre delle figure.
- Cancella tutte le variabili nell'area di lavoro.
- Cancella la finestra di comando.
- Visualizzazione nella finestra di comando: inserire le seguenti dimensioni per le scansioni MRI:
- Su una nuova riga nella finestra di comando, chiedi: Lunghezza di una scansione in millimetri:. La risposta inserita dall'utente sarà impostata sulla variabile "lengthMM".
- Su una nuova riga, chiedi: Distanza tra le scansioni MRI in millimetri:. La risposta inserita dall'utente verrà impostata sulla variabile "ZStacks".
Passaggio 15: Il codice principale: elaborazione in batch delle immagini

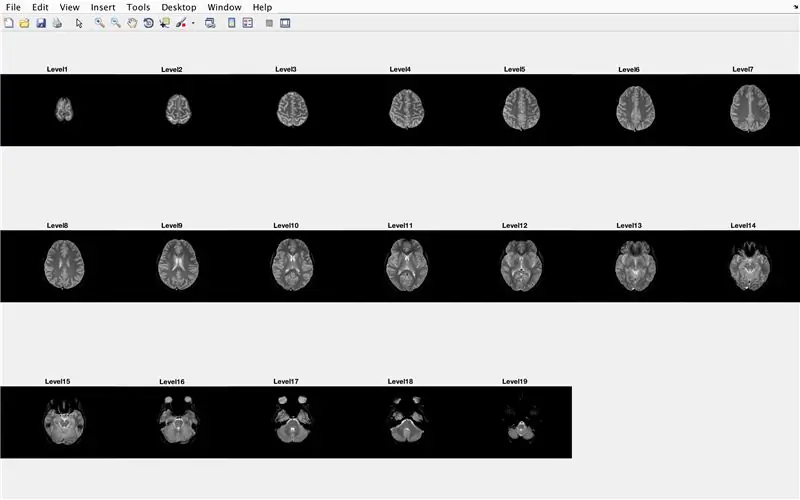
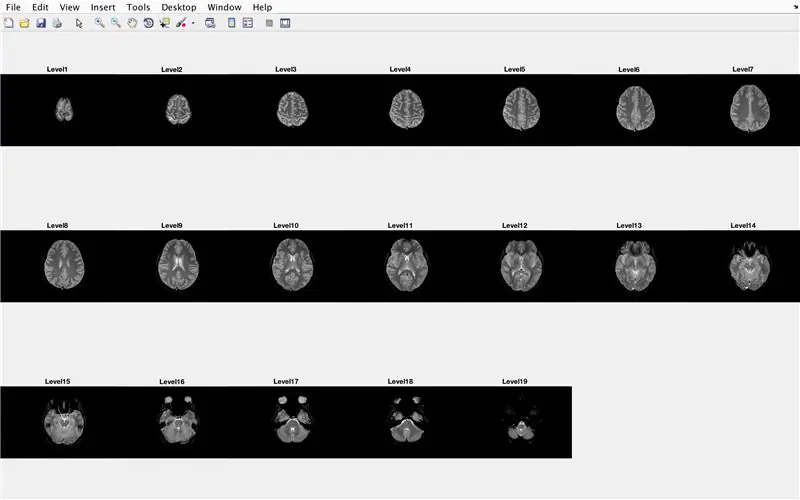
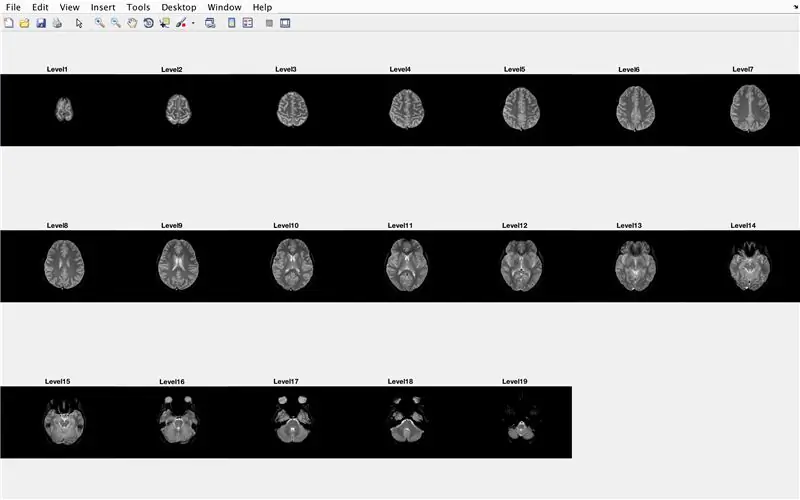
In questa sezione, il codice caricherà le immagini (costituite dalle scansioni MRI delle sezioni trasversali del cervello) e memorizzerà i nomi di ciascun file immagine nella variabile "Base" e visualizzerà ciascuna delle scansioni MRI. Si prega di seguire con il codice nell'immagine, che fa quanto segue:
- Crea l'array della struttura "BrainImages" che contiene informazioni su tutti i file all'interno della cartella corrente che si adattano al formato del nome di MRI_().png
- Imposta la variabile "NumberofImages" uguale al numero di elementi nell'array della struttura "BrainImages"
- Apri la finestra della figura 1
- Imposta un ciclo for per scorrere il numero di immagini conteggiate nel file
- Per ogni ciclo, "CurrentImage" è il rispettivo nome di ogni file MRI_i.png, con il numero di iterazione come 'i'
- Genera una sottotrama 3 x 7 per visualizzare le 19 immagini da caricare da "imshow"
- Visualizza ogni immagine come un altro elemento nella finestra della figura della sottotrama
- Intitola ogni elemento della sottotrama come Livello_, dove vuoto è il numero di iterazione del ciclo for.
- Termina il ciclo for (evitando errori)
Questo mostrerà nella finestra di figura 1 tutte le scansioni MRI in forma grezza in una configurazione 3 x 7 senza spazi nell'orientamento x.
Passaggio 16: Il codice principale: Imbottitura

Con l'imbottitura, evitiamo il problema di leggere discrepanze nelle dimensioni dell'immagine che potrebbero generare errori per mancata corrispondenza delle dimensioni nel caso in cui un'immagine sia leggermente più grande di un'altra.
- Apri la finestra della figura 2
- Carica la matrice dell'immagine da MRI_1-p.webp" />
- Trova la dimensione della matrice dell'immagine e imposta su "OriginalXPixels" (per numero di righe) e "OriginalYPixels" (per numero di colonne)
- Imposta la matrice "BrainMat" in modo che sia composta da tutti gli zeri con 20 righe in più e 20 colonne in più per ogni piano e 19 sezioni trasversali totali, una per piano.
- Imposta "HolesMat" in modo che contenga lo stesso array tridimensionale di zeri per inserire le coordinate del foro in un secondo momento
- Crea "zeroMat" in modo che sia la dimensione del pad più venti righe e venti colonne, una matrice bidimensionale di zeri.
Passaggio 17: Il codice principale: determinazione dei confini
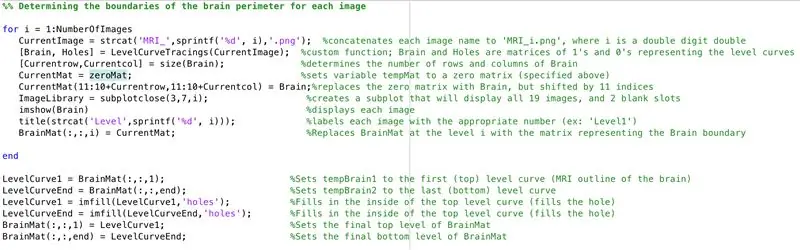
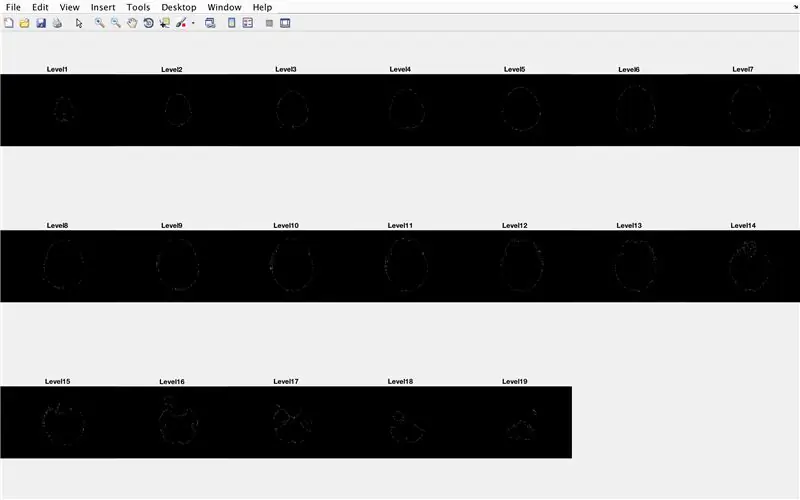
- Imposta un ciclo for per esaminare i dati di ogni immagine caricata in precedenza
- Allo stesso modo dell'elaborazione batch in precedenza, "CurrentImage" carica i file con "MRI_i.png", dove i è il numero di iterazione
- Esegui ogni immagine attraverso la funzione di elaborazione "LevelCurveTracings2.m" che hai creato in precedenza
- Trova la dimensione dell'output "Brain", imposta il numero di righe su "Currentrow" e il numero di colonne su "Currentcolumns"
- Imposta "CurrentMat" su una matrice di zeri con le dimensioni specificate da "Currentrow" e "Currentcolumns"
- Centrare i dati da "Brain" in "CurrentMat", con un margine di 10 righe su tutti i lati
- Genera una sottotrama di dimensioni 3 x 7, per visualizzare i confini delle immagini
- Dai un titolo a ciascuno degli elementi della sottotrama nella finestra della figura
- Genera matrice tridimensionale "BrainMat" composta da ogni strato di limiti "CurrentMat"
- Termina il ciclo for (per evitare errori)
La sottosezione seguente riempie i fori lasciati nella parte superiore e inferiore della forma tridimensionale proposta
- Imposta "LevelCurve1" uguale al primo strato di "BrainMat" (fondo del solido)
- Imposta "LevelCurveEnd" uguale allo strato finale di "BrainMat" (parte superiore del solido)
- Sovrascrivi "LevelCurve1" con un livello riempito
- Sovrascrivi "LevelCurveEnd" con un livello riempito
- Imposta il livello riempito come livello inferiore di "BrainMat"
- Imposta il livello riempito come livello superiore di "BrainMat"
Passaggio 18: Il codice principale: determinare la corretta dimensione Z

Le prime tre righe consistono nell'impostare un array vuoto "z", e nel fare semplici operazioni di conversione (dividere i pixel per lunghezza) per ottenere una corretta lettura del volume in mm^3
- Crea un ciclo for per scorrere ogni livello
- Trova il numero di unità in un dato livello
- Converti le coordinate z per quelle in valori che vengono ridimensionati secondo il rapporto corretto, impostato su "tempz", un vettore colonna
- Aggiungi il valore z per la curva di livello al vettore z
Con questo le coordinate z vengono regolate correttamente.
Passaggio 19: Il codice principale: determinazione delle coordinate X e Y

Ora per determinare le posizioni xey di ciascuno dei punti nei confini.
- Inizializza "xBrain" come un array vuoto
- Inizializza "yBrain" come un array vuoto
- Imposta un ciclo for per scorrere ogni immagine caricata
- Compila una matrice a due colonne per memorizzare le coordinate planari di ciascun punto nel limite, rappresentato dai vettori colonna "RowBrain" e "ColumnBrain"
- Aggiungi "xBrain" con le coordinate "RowBrain" attualmente trovate
- Aggiungi "yBrain" con le coordinate "ColumnBrain" attualmente trovate
- Termina il ciclo for (per evitare errori)
Passaggio 20: Il codice principale: tracciare una struttura tridimensionale, trovare il volume e registrare i dati
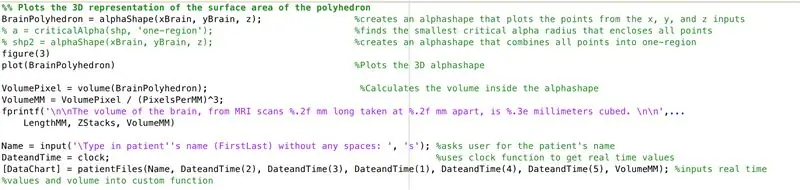
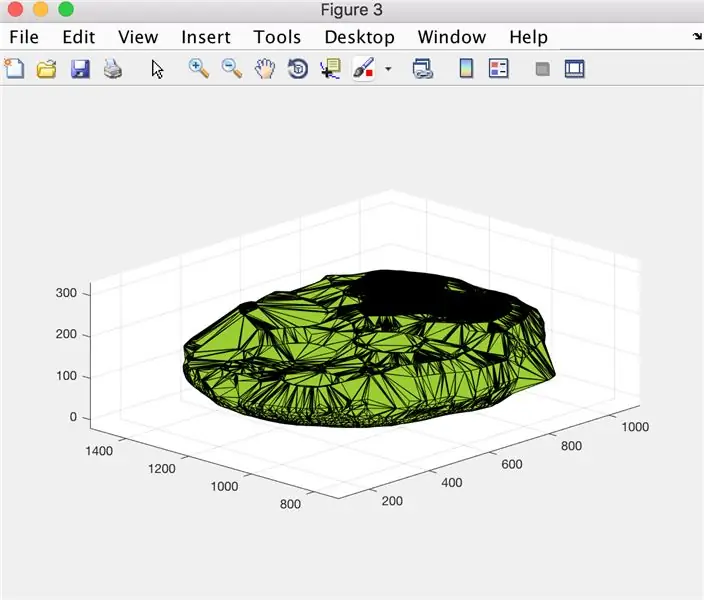

Usando la funzione alphaShape, creeremo una struttura tridimensionale da cui possiamo calcolare il volume del cervello.
- Usa la funzione alphaShape, collega i vettori "xBrain", "yBrain" e "z" per le coordinate x, yez e imposta uguale a "BrainPolyhedron"
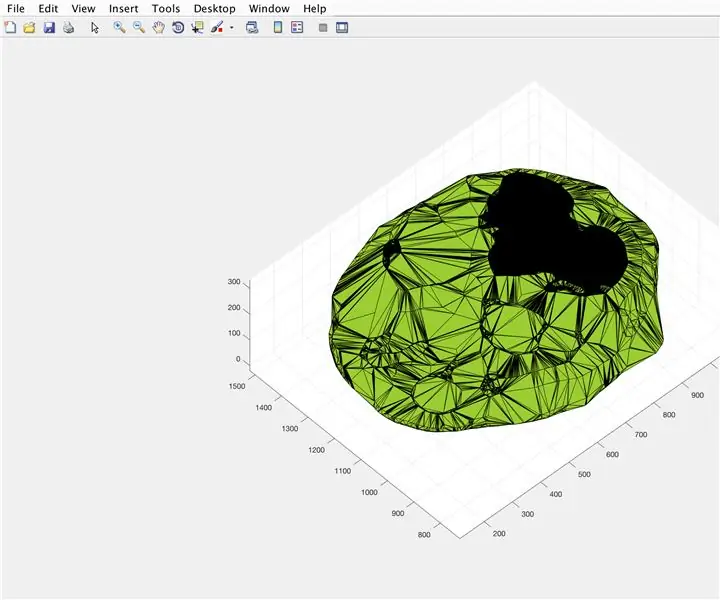
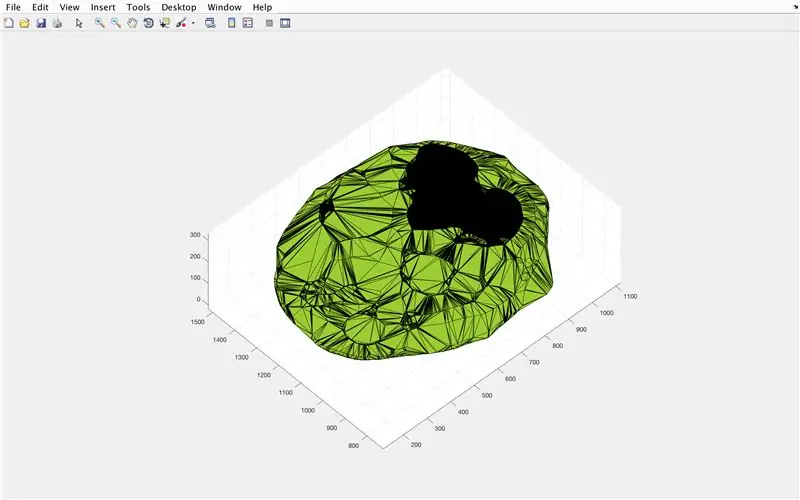
- Apri la finestra della figura 3
- Traccia la forma alfa calcolata "BrainPolyhedron", visualizza nella finestra della figura
- Calcola il volume della forma alfa, usando una funzione "volume" che funziona per le forme alfa
- Converti il volume in mm^3
- Stampa il volume del solido nella finestra di comando
- Richiedi di impostare un nome paziente come input
- Ottieni la data e l'ora correnti con l'orologio e imposta su "DateandTime"
- Richiama la funzione "filepaziente" per registrare e tracciare i dati calcolati
Da qui la seconda e la terza immagine dovrebbero mostrare le figure che compaiono e la quarta immagine quella che dovrebbe essere visualizzata nella finestra di comando.
Consigliato:
Trasforma un telefono rotante in una radio e viaggia nel tempo: 5 passaggi (con immagini)

Trasforma un telefono rotante in una radio e viaggia nel tempo: ho hackerato un telefono rotante in una radio! Prendi il telefono, scegli un paese e un decennio e ascolta dell'ottima musica!Come funzionaQuesto telefono rotante ha un microcomputer integrato (un Raspberry Pi), che comunica con radiooooo.com, una web radio. Il
Sistema di monitoraggio intelligente del tempo e della velocità del vento basato su IOT: 8 passaggi

Sistema di monitoraggio intelligente del tempo e della velocità del vento basato su IOT: sviluppato da - Nikhil Chudasma, Dhanashri Mudliar e Ashita RajIntroduzione L'importanza del monitoraggio del tempo esiste in molti modi. I parametri meteorologici devono essere monitorati per sostenere lo sviluppo in agricoltura, serra
Kit per auto di monitoraggio robot intelligente fai-da-te Monitoraggio fotosensibile dell'auto: 7 passaggi

Fai da te Smart Robot Tracking Car Kit Tracking Car Photosensitive: Design by SINONING ROBOTÈ possibile acquistare da tracking robot carTheoryLM393 chip confrontare i due fotoresistori, quando c'è un LED fotoresistore laterale su BIANCO il lato del motore si fermerà immediatamente, l'altro lato del motore girare, in modo che
Monitoraggio del tempo utilizzando ESP32_DHT11_OLED_Thingspeak: 7 passaggi

Monitoraggio meteo utilizzando ESP32_DHT11_OLED_Thingspeak: In questo tutorial, creerai un monitor meteo che monitora la temperatura e l'umidità, utilizzando un ESP32 e DHT11, viene visualizzato su un display OLED. Ed è caricato su ThingSpeak. L'ESP32 è un potente strumento IOT. È un sistema a basso costo
Monitoraggio in tempo reale del valore del tuo sensore da qualsiasi parte del mondo: 4 passaggi

Monitoraggio in tempo reale del valore del tuo sensore da qualsiasi parte del mondo: ho ricevuto un messaggio sul numero WhatsApp di techiesms relativo all'aiuto per la realizzazione di un progetto. Il progetto consisteva nel misurare la pressione esercitata sul sensore di pressione e visualizzarla su smartphone. Così ho aiutato a realizzare quel progetto e ho deciso di fare un tutor
