Sommario:
- Passaggio 1: prerequisiti
- Passaggio 2: prerequisiti (continua)
- Passaggio 3: prerequisiti (continua)
- Passaggio 4: prerequisiti (continua)
- Passaggio 5: ripulire Matlab per prepararsi all'esecuzione del codice
- Passaggio 6: selezionare 10 immagini dell'occhio normale e 10 immagini con sintomi di retinopatia diabetica
- Passaggio 7: selezionare 10 immagini di occhi normali e 10 immagini con sintomi di retinopatia diabetica (continua)
- Passaggio 8: creare 2 variabili (normale e diagnosticata) e impostarle ciascuna uguale a 0
- Passaggio 9: creare un ciclo for per caricare automaticamente immagini normali
- Passaggio 10: creare un ciclo for per caricare automaticamente immagini normali (continua)
- Passaggio 11: ritaglia i bordi dell'immagine
- Passaggio 12: creare un'immagine in scala di grigi
- Passaggio 13: crea un'immagine a contrasto
- Passaggio 14: Migliora l'immagine a contrasto
- Passaggio 15: creare un filtro medio
- Passaggio 16: combinare il filtro della media con l'immagine contrastata
- Passaggio 17: crea una nuova maschera media sottraendo pixel
- Passaggio 18: creare un'immagine filtrata binaria
- Passaggio 19: rimuovere i BLOB più piccoli trovati nelle immagini filtrate
- Passaggio 20: creare un elemento di struttura del disco
- Passaggio 21: eseguire operazioni di chiusura morfologica
- Passaggio 22: trova gli oggetti con connettività di almeno 8
- Passaggio 23: trova il numero massimo di pixel collegati
- Passaggio 24: imposta i valori massimi dei pixel su 0 e trova i pixel con connettività >=26 pixel
- Passaggio 25: rimuovere i vasi sanguigni nell'immagine
- Passaggio 26: visualizzazione della figura
- Passaggio 27: rimuovere i vasi e contare le macchie di sangue
- Passaggio 28: diagnosticare l'immagine retinica in base al numero di coaguli di sangue identificati
- Passaggio 29: se sono presenti più di 5 BLOB…
- Passaggio 30: ripetere il processo di filtraggio per immagini normali con valori delle cifre dell'immagine come 2 e 3
- Passaggio 31: ripetere l'intero processo per le immagini diagnosticate
- Passaggio 32: analisi statistica
- Passaggio 33: trovare l'intervallo di confidenza
- Autore John Day day@howwhatproduce.com.
- Public 2024-01-30 10:04.
- Ultima modifica 2025-01-23 14:50.

(Vedi schema del codice sopra)
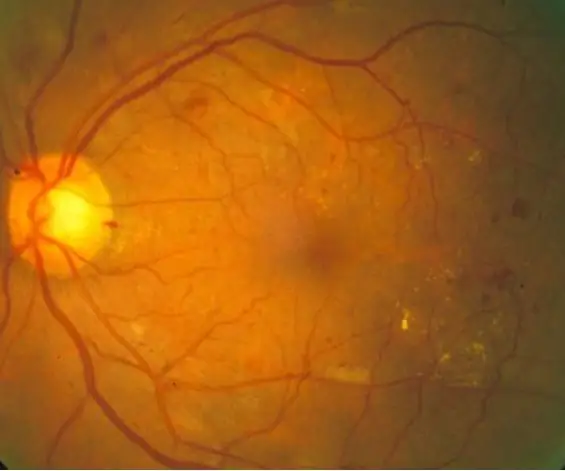
La retinopatia diabetica è una malattia oculare correlata al diabete causata da alti livelli di zucchero nel sangue. Gli alti livelli di zucchero nel sangue fanno gonfiare i vasi sanguigni nelle retine, il che porta a vasi sanguigni dilatati e persino perdite di vasi, che portano a macchie scure nelle immagini retiniche. Con questo codice, miriamo a utilizzare l'aspetto delle macchie di perdita dei vasi sanguigni come indicatore di retinopatia diabetica di fondo, anche se nel mondo reale sarebbero necessarie ulteriori tecniche di diagnosi. L'obiettivo di questo codice è automatizzare l'elaborazione delle immagini e la diagnosi delle immagini retiniche per identificare i segni di retinopatia diabetica mostrati attraverso macchie scure nelle immagini retiniche.
10 immagini retiniche normali e 10 immagini retiniche diagnosticate sono state elaborate attraverso un codice che prima legge e filtra le immagini e poi quantifica le macchie scure per determinare se sono presenti sintomi di retinopatia diabetica, in base a una determinata soglia. I risultati vengono quindi stampati nella finestra di comando per l'interpretazione del visualizzatore.
Passaggio 1: prerequisiti
1. Assicurati di aver scaricato il programma MATLAB sul tuo computer.
2. Scarica il file txt che trovi nel link. (Premi "ctrl+s" per salvare nella stessa directory del codice MATLAB)
Passaggio 2: prerequisiti (continua)
4. Apri MATLAB e digita "uiimport" nella finestra di comando.
5. Seleziona il file officialdiagnoses.txt e importalo in MATLAB come matrice di celle.
6. Assicurati di vedere "diagnosi ufficiali" come variabile nell'area di lavoro.
Passaggio 3: prerequisiti (continua)

7. Scarica la funzione ModWald.m, che puoi ottenere dal codice sopra o scaricalo da Canvas.
(Codice fornito dal Professor King e dal Professor Choi)
Passaggio 4: prerequisiti (continua)

8. Scarica le 400 immagini grezze dalla sezione dati di The STARE Project.
Passaggio 5: ripulire Matlab per prepararsi all'esecuzione del codice

Aggiungi al codice:
1. chiudi tutto (chiude tutte le immagini aperte in precedenza)
2. clearvars - tranne le diagnosi ufficiali (cancella tutte le variabili tranne il file txt delle diagnosi ufficiali precedentemente importato)
3. cclc (cancella la finestra di comando)
Passaggio 6: selezionare 10 immagini dell'occhio normale e 10 immagini con sintomi di retinopatia diabetica
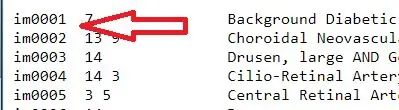
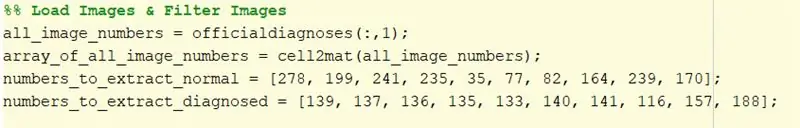
1. Prendere il file di testo della diagnosi ed estrarre i nomi delle immagini. Questi nomi sono contenuti nella prima colonna del file di testo quindi per estrarli digita 'officialdiagnoses(:, 1)'. La matrice dei nomi delle immagini è stata assegnata a una variabile, "all_image_numbers"
2. Converti la variabile all_image_numbers da un array di celle a un array di matrici utilizzando la funzione cell2mat
Passaggio 7: selezionare 10 immagini di occhi normali e 10 immagini con sintomi di retinopatia diabetica (continua)
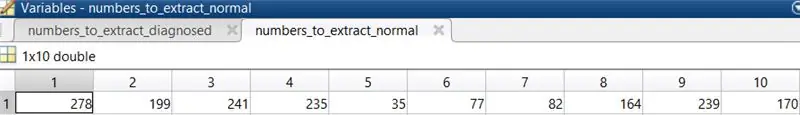
3. Selezionare 10 immagini dell'occhio normale per eseguire il codice. Le immagini selezionate in questo caso erano 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Metti questi numeri in una matrice e assegnali a una variabile che verrà chiamata durante il caricamento delle immagini.
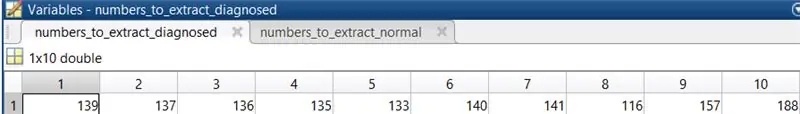
4. Ripetere il passaggio 3 per le immagini retiniche a cui è stata diagnosticata la retinopatia diabetica. Le immagini selezionate in questo caso erano 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Passaggio 8: creare 2 variabili (normale e diagnosticata) e impostarle ciascuna uguale a 0

Crea queste variabili prima del ciclo for per inizializzare i numeri del ciclo.
Passaggio 9: creare un ciclo for per caricare automaticamente immagini normali

1. Crea un ciclo for
2. Impostare una variabile di conteggio (i, in questo caso) su una matrice di valori 1-10. Questa variabile di conteggio verrà utilizzata per chiamare ogni immagine individualmente
3. Prendi l'elemento i nella matrice di immagini per estrarre e convertire il nome dell'immagine da una stringa a un numero utilizzando la funzione num2str.
Trova il numero di cifre presenti nel nome dell'immagine utilizzando la funzione numel. Assegna questo valore a una variabile, digits_normal. Questo numero deve essere 1 per i numeri a una cifra, 2 per i numeri a due cifre e 3 per i numeri a tre cifre. Queste informazioni verranno utilizzate per chiamare automaticamente le immagini.
Passaggio 10: creare un ciclo for per caricare automaticamente immagini normali (continua)

3. Creare un'istruzione if contenente tutte e tre le possibilità dei passaggi precedenti. Se il nome dell'immagine ha 1 cifra, l'immagine sarà chiamata "im000", se ha 2 cifre, l'immagine sarà chiamata "im00", e se ha 3 l'immagine sarà chiamata "im0".
4. Sotto ogni istruzione if, assegna una variabile a imread “im” sotto la corrispondente istruzione if con il numero appropriato di zeri (come descritto sopra), seguito da i.
Passaggio 11: ritaglia i bordi dell'immagine
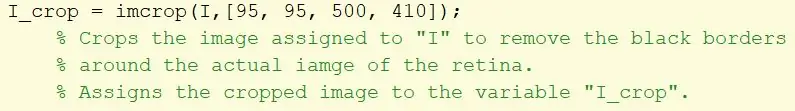
Prendi l'immagine originale e applica un filtro imcrop per eliminare i bordi neri e assegnarlo a una variabile I_crop. Il rettangolo di ritaglio viene specificato utilizzando una matrice [95, 95, 500, 410].
Passaggio 12: creare un'immagine in scala di grigi

Prendi l'immagine ritagliata e applica il filtro rbg2gray per cambiare l'immagine in scala di grigi. Assegna questa immagine alla variabile I2.
Passaggio 13: crea un'immagine a contrasto
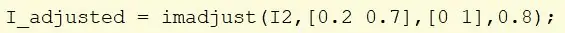
Prendi l'immagine I2 e usa imadjust per ridimensionare i valori di intensità.
Prendi i valori che rientrano nell'intervallo [0,2, 0,7] e ridimensionali a [0, 1]. La gamma è impostata su 0,8 per rendere l'immagine più luminosa. Assegna la nuova immagine a I_adjusted.
Passaggio 14: Migliora l'immagine a contrasto
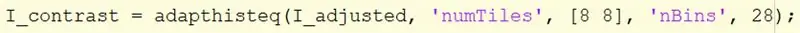
Prendi l'immagine I_adjusted e usa la funzione Adapthisteq per migliorare il contrasto.
La sintassi Adapthisteq richiede il nome dell'immagine, I_adjusted, "numTiles", la dimensione di numTiles, "nBins" e il numero di bin. La dimensione di numTiles è impostata su [8 8], dividendo l'immagine in tile 8x8 e il numero di bin è impostato su 28. Assegna l'immagine a I_constrast.
Passaggio 15: creare un filtro medio

Crea una variabile chiamata 'meanfilt' usando la funzione fspecial. Immettere la "funzione media" per creare il filtro della media e inserire [90 90] per la dimensione della finestra scorrevole.
Passaggio 16: combinare il filtro della media con l'immagine contrastata
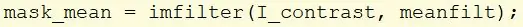
Crea una nuova variabile denominata mask_mean e usa la funzione imfilter per prendere l'immagine I_contrast e applicare il filtro medio creato in precedenza.
Passaggio 17: crea una nuova maschera media sottraendo pixel

Crea una variabile denominata mask_mean2 e usa la funzione imsubtract per sottrarre il valore di ogni pixel in I_contrast dal pixel corrispondente in mask_mean.
Passaggio 18: creare un'immagine filtrata binaria

Trasforma le immagini in scala di grigi in bianco e nero usando imbinarize. Input mask_mean2, 'adaptive', 'ForegroundPolarity', 'dark', 'Sensitivity', 0.6. Assegna questa nuova immagine a mask_binarize.
Passaggio 19: rimuovere i BLOB più piccoli trovati nelle immagini filtrate
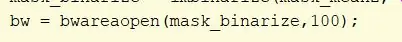
Rimuovi gli oggetti con connettività inferiore a 100 pixel utilizzando la funzione bwareaopen su mask_binarize e imposta il valore di soglia su 100. Assegna la variabile come bw.
Passaggio 20: creare un elemento di struttura del disco

Creare un elemento strutturante del disco (con raggio 2) utilizzando la funzione strel. Assegnalo a se.
Passaggio 21: eseguire operazioni di chiusura morfologica

Prendi bw e applica la funzione di chiusura all'elemento strutturale per eseguire un'operazione di chiusura morfologica sull'oggetto.
Passaggio 22: trova gli oggetti con connettività di almeno 8
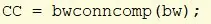
Prendi bw e usa bwconncomp per trovare gli oggetti con connettività di almeno 8 nell'immagine. Assegnare l'output numerico a cc_1.
Passaggio 23: trova il numero massimo di pixel collegati
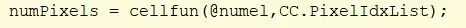
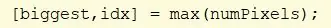
Usa la funzione cellfun per eseguire la funzione "numel" su ogni cella in CC. Questo trova il numero di elementi nella cella PixelIdxList. Assegna valore a "numPixels".
Trova i valori massimi in numPIxels. Assegna il massimo più grande a "più grande" e l'indice del valore massimo a "idx".
Passaggio 24: imposta i valori massimi dei pixel su 0 e trova i pixel con connettività >=26 pixel
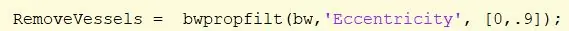



Imposta i pixel con i valori più grandi nell'immagine "bw" su 0, rendendo i pixel neri.
Trova gli oggetti con una connettività di almeno 26 pixel nell'immagine usando bwconncomp. Assegna alla variabile cc_1.
Passaggio 25: rimuovere i vasi sanguigni nell'immagine
Rimuovere i vasi sanguigni ancora presenti nell'immagine utilizzando la funzione bwpropfilt con un intervallo di [0, 0,9].
[0.9, 1] è escluso perché i valori prossimi a 1 indicano una linea. Assegna a "Rimuovi vasi".
Passaggio 26: visualizzazione della figura
Visualizza ogni immagine filtrata in una sottotrama. Imshow. con input "border" e "tight", visualizza ogni immagine in una struttura di sottotrama. Aggiungi un titolo a ciascuna immagine per distinguere quale filtro è stato utilizzato.
Passaggio 27: rimuovere i vasi e contare le macchie di sangue
1. Prendi "RemoveVessels" e applica la funzione "Centroid" in regionprops per identificare i centroidi degli oggetti nell'immagine. Questi oggetti dovrebbero corrispondere ai coaguli di sangue presenti nell'immagine.
2. Contare il numero di coaguli di sangue identificati prendendo la lunghezza della matrice centroide.
Passaggio 28: diagnosticare l'immagine retinica in base al numero di coaguli di sangue identificati
Utilizzare le istruzioni if per diagnosticare l'immagine in base al numero di coaguli di sangue identificati.
Se il numero di centroidi identificati era inferiore o uguale a 5, l'immagine veniva identificata come normale.
Se il numero di centroidi era maggiore di 5, l'immagine veniva diagnosticata con retinopatia diabetica.
Il risultato viene stampato nella finestra di comando utilizzando fprintf.
Passaggio 29: se sono presenti più di 5 BLOB…
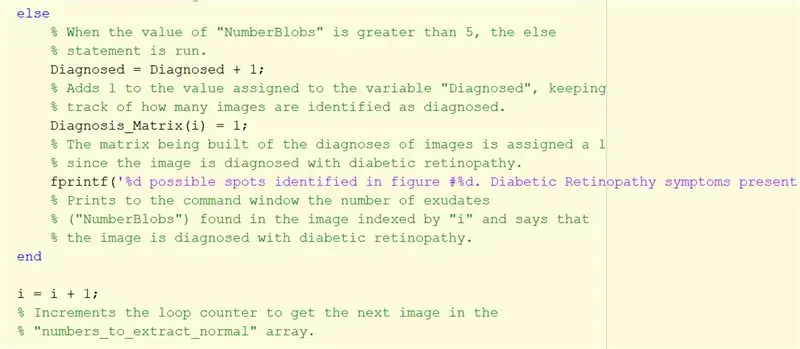
Ripetere le istruzioni precedenti per le immagini diagnosticate come istruzione else. Questa parte verrà eseguita se il numero di blob è maggiore di 5.
Termina l'istruzione if.
Passaggio 30: ripetere il processo di filtraggio per immagini normali con valori delle cifre dell'immagine come 2 e 3
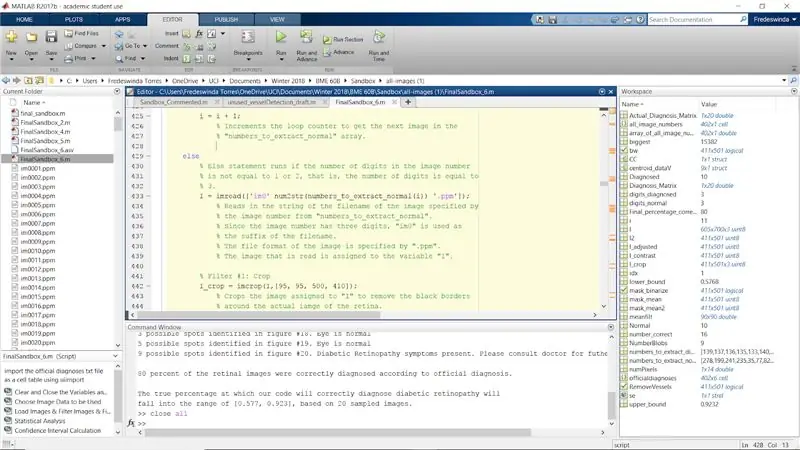
Ripetere il processo per il resto dell'istruzione if originale quando numel (il numero di cifre nel numero dell'immagine) è uguale a 2 e 3. Questo completa il ciclo for per le immagini normali.
Termina il ciclo for.
Passaggio 31: ripetere l'intero processo per le immagini diagnosticate
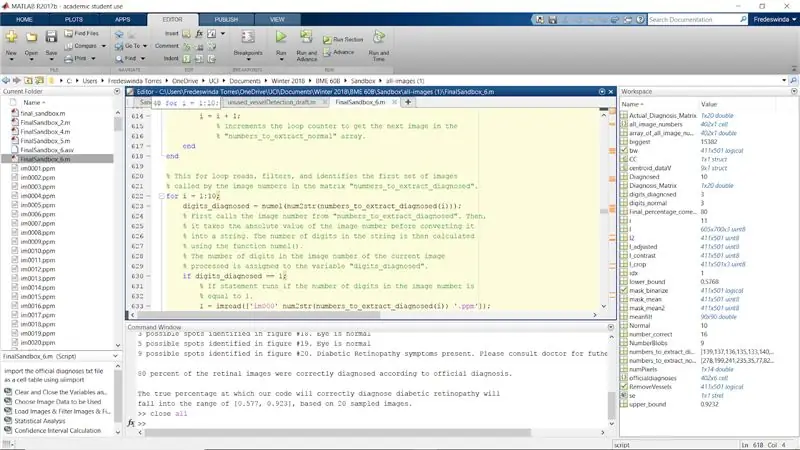
Ripetere l'intero processo utilizzando le immagini diagnosticate elencate dalla matrice "numbers_to_extract_diagnosed".
Assicurati di esaminare ogni cifra (i) e cambiarla in figura (i+10) in modo che le cifre diagnosticate compaiano come immagini da 11 a 20.
Passaggio 32: analisi statistica
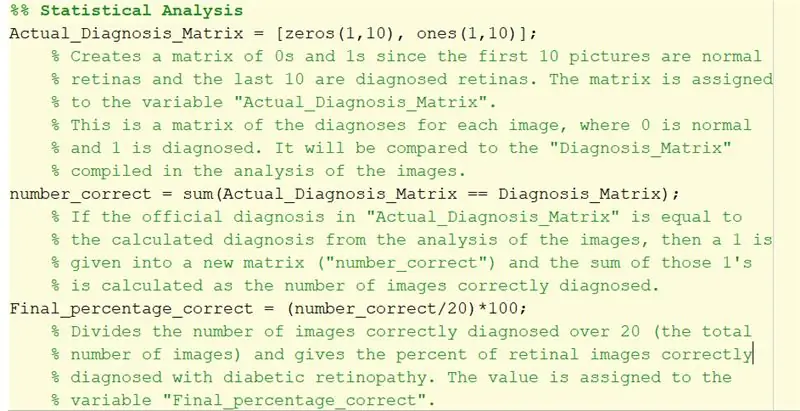
1. 'Actual_Diagnosis_Matrix' viene utilizzato per confrontare i risultati con la diagnosi ufficiale trovata nel file txt. I primi 10 zeri indicano che le prime 10 immagini dovrebbero essere normali. Le ultime 10 indicano che le ultime 10 immagini dovrebbero essere classificate come retinopatia diabetica.
2. Il doppio segno di uguale utilizzato per creare 'number_correct' crea un array logico confrontando il valore degli elementi corrispondenti di 'Actual_Diagnosis_Matrix' con 'Diagnosis_Matrix' creato dal ciclo for.
Per ogni elemento che corrisponde alla diagnosi verrà aggiunto un 1, che indica il codice correttamente diagnosticato quell'immagine. Se non è corretto aggiungerà uno 0 alla matrice.
Quindi, prendendo la somma di ciò, si sommano tutti. In altre parole, trova la somma delle immagini correttamente diagnosticate.
3. 'Final_percentage_correct' è la percentuale calcolata dell'accuratezza con cui il codice ha diagnosticato la retinopatia diabetica. Il numero di immagini diagnosticate correttamente viene diviso per 20 (il numero totale di immagini) e moltiplicato per 100 per trovare la percentuale di diagnosi riuscite.
Passaggio 33: trovare l'intervallo di confidenza
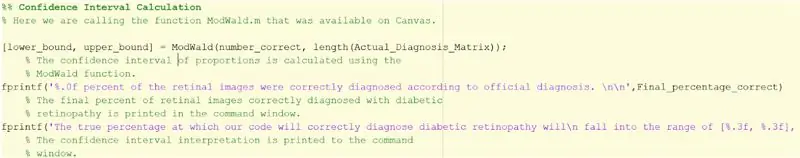
1. Assicurati di aver scaricato ModWald.m per chiamarlo come funzione. Senza la funzione, dovresti calcolare tu stesso l'intervallo di confidenza utilizzando il metodo Wald modificato.
2. La funzione ModWald dispone di 2 ingressi dove il primo è il numero di immagini correttamente identificate e il secondo è la quantità totale di immagini.
3. La funzione ModWald restituirà i limiti inferiore e superiore dell'intervallo di confidenza delle proporzioni per l'accuratezza dei dati campionati. In altre parole, fornirà un intervallo di percentuali in cui risiederà la vera percentuale di accuratezza del codice.
4. Utilizzare fprintf di seguito per visualizzare le statistiche e l'intervallo di confidenza nella finestra di comando.
> fprintf('%.0f percento delle immagini retiniche sono state correttamente diagnosticate secondo la diagnosi ufficiale. \n\n', Final_percentage_correct)
> fprintf('La vera percentuale alla quale il nostro codice diagnosticherà correttamente la retinopatia diabetica\n rientrerà nell'intervallo di [%.3f, %.3f], basato su 20 immagini campionate \n', lower_bound, upper_bound)
Consigliato:
Macchina per lo stampaggio a iniezione automatizzata per il riciclaggio della plastica: 5 passaggi

Macchina per lo stampaggio a iniezione automatizzata per il riciclaggio della plastica: Ciao :) Questo Instructable riguarda la nostra "macchina per lo stampaggio a iniezione automatizzata per il riciclaggio della plastica". (chiamato: Smart Injector) L'idea alla base della macchina è quella di offrire una soluzione decentralizzata di riciclaggio della plastica. Il riciclaggio è spesso limitato
Assistenza al parcheggio Facile riparazione / diagnosi: 4 passaggi

Assistenza al parcheggio Facile riparazione / diagnosi: Ok, iniziamo, ho una Chevrolet Avalanche del 2010 e ha 4 sensori di assistenza al parcheggio nel paraurti posteriore. Questo intrattabile può essere utilizzato su e veicolo al meglio delle mie conoscenze, se hai davanti o Rea o entrambi. Così sono andato al mio preferito
Dischi rigidi: diagnosi, risoluzione dei problemi e manutenzione: 3 passaggi

Dischi rigidi: diagnosi, risoluzione dei problemi e manutenzione: cos'è un disco rigido? - In parole povere, il disco rigido è ciò che memorizza tutti i tuoi dati. Ospita il disco rigido, dove si trovano fisicamente tutti i file e le cartelle. Le informazioni sono memorizzate magneticamente sul disco, quindi rimangono sull'unità anche quando
MESOMIX - Macchina per la miscelazione della vernice automatizzata: 21 passaggi (con immagini)

MESOMIX - Macchina per miscelare la vernice automatizzata: sei un designer, un artista o una persona creativa che ama gettare i colori sulla tua tela, ma spesso è una lotta quando si tratta di creare la tonalità desiderata. Quindi, questa istruzione tecnica artistica svanirà che lottano nel nulla. Poiché questo dispositivo, tu
Guida alla codifica/diagnosi BMW INPA E60: 4 passaggi

Guida alla codifica/diagnosi BMW INPA E60: in questo articolo raccoglierò alcuni casi sul caso di lavoro BMW INPA su BMW E60 e se hai altri casi BMW INPA E60 che desideri condividere con le persone, ti preghiamo di inviarlo alla mia e-mail. lo metterò qui per condividerlo
