Sommario:
- Passaggio 1: avviare l'interfaccia utente grafica (GUI)
- Passaggio 2: caricamento e lettura di immagini MRI in MATLAB
- Passaggio 3: filtraggio delle immagini
- Passaggio 4: isolamento del tumore tramite maschera ellittica
- Passaggio 5: delineazione del tumore
- Passaggio 6: analisi delle proprietà fisiche dei tumori
- Autore John Day day@howwhatproduce.com.
- Public 2024-01-30 10:02.
- Ultima modifica 2025-01-23 14:49.
Di: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Questo codice MATLAB è un programma per rilevare l'esatta dimensione, forma e posizione di un tumore trovato nelle scansioni MRI del cervello di un paziente. Questo programma è progettato per funzionare originariamente con il rilevamento del tumore nelle scansioni MRI del cervello, ma può anche essere utilizzato per la diagnostica del cancro anche in altre scansioni di organi.
Le seguenti istruzioni descriveranno prima i metodi per l'analisi delle immagini attraverso il filtraggio e la pulizia della scansione MRI, attraverso la binarizzazione, il filtraggio mediano e le finestre scorrevoli. Successivamente, indicherà come isolare il tumore utilizzando una maschera ellittica pre-generata e filtrandola ulteriormente per delineare il perimetro della forma del tumore.
Una volta rilevato il tumore, le istruzioni descriveranno ulteriormente come incorporare questo programma in un'interfaccia utente grafica (GUI). In queste istruzioni, verranno allegati il codice e i file appropriati per aiutare a spiegare come funziona questa analisi della scansione MRI.
Alcune cose da sapere, scaricare e avere a disposizione prima di procedere con questa istruzione:1. Assicurati di aver scaricato l'ultima versione di MATLAB. Puoi installare R2018b qui:
2. Per eseguire questo programma, è necessario avere accesso ai file di scansione cerebrale MRI. Sebbene alcuni possano sempre essere trovati dalle immagini di Google, è possibile eseguire un'analisi approfondita e accurata da immagini corrette di vari strati di scansioni cerebrali per ciascun paziente. È possibile accedere ai file per 20 diversi pazienti con glioblastoma pre e post trattamento da questo database:
3. Il focus di questo programma e i vari metodi che guidano questo progetto sono delineati in questo documento di ricerca:
Passaggio 1: avviare l'interfaccia utente grafica (GUI)
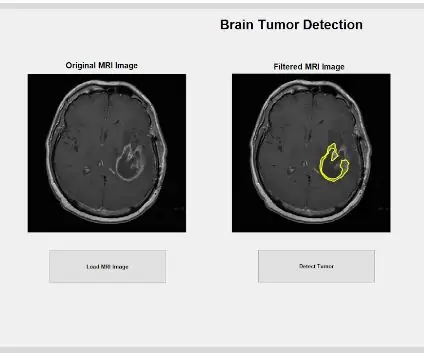
Il primo passo sarebbe creare e avviare l'interfaccia utente grafica, GUI. Questo può essere fatto digitando guide nella finestra di comando, premendo invio e creando una nuova GUI. Una volta completato questo passaggio, puoi iniziare a creare funzioni come assi, testo statico, modifica testo e pulsanti che verranno visualizzati una volta eseguito il programma e con cui l'utente potrà interagire. Queste funzioni possono essere modificate e manipolate tramite la finestra di ispezione delle proprietà, tuttavia la caratteristica più importante che deve essere modificata durante la creazione di queste funzioni è il nome del tag. È importante modificare il nome del tag di ogni funzione implementata perché ci consentirà di creare una funzione di callback distinguibile. Una volta che sei soddisfatto del layout della tua GUI, puoi passare al caricamento dei file DICOM che verranno visualizzati all'interno della GUI.
Passaggio 2: caricamento e lettura di immagini MRI in MATLAB
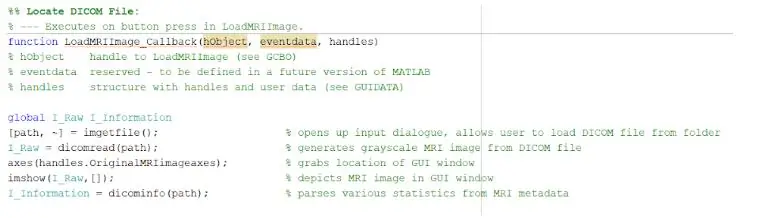
Per caricare i file DICOM dovresti inizializzare correttamente la funzione di richiamata che verrebbe eseguita quando si preme il pulsante "Carica immagine MRI". Una volta completata questa operazione, è necessario creare una variabile globale che visualizzi l'immagine sugli assi delle maniglie in cui si desidera visualizzare l'immagine MRI originale. Le immagini della scansione MRI scaricate dal database sono tutti file in formato DICOM che devono essere caricati nella directory MATLAB. Individua il file utilizzando imgetfile per caricarli nel programma. Le immagini vengono lette utilizzando la funzione MATLAB incorporata "dicomread" e la prima immagine non elaborata per ciascun file viene incorporata negli assi della GUI di sinistra utilizzando imshow.
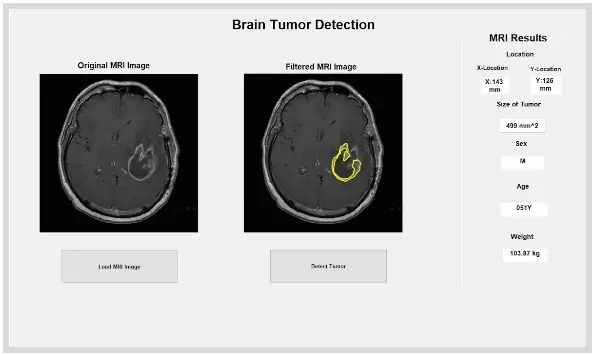
Anche la funzione MATLAB integrata "dicominfo" è estremamente utile per indirizzare tutte le informazioni di ciascun file dicom MRI. Abbiamo utilizzato questa funzione per estrarre tutte le informazioni descrittive dei pazienti, come sesso, età, peso e altezza. Questa funzione fornisce anche l'ordine dello stack utile per l'implementazione del programma all'interno dell'interfaccia utente grafica. Abbiamo creato variabili per ciascuna delle informazioni descrittive dei pazienti che verranno utilizzate per la GUI quando viene premuto il pulsante di rilevamento.
Passaggio 3: filtraggio delle immagini

Una volta che il file DICOM dell'immagine grezza è stato caricato e letto, l'immagine deve essere convertita da scala di grigi in una forma binarizzata composta da soli pixel in bianco e nero Abbiamo usato la funzione 'imbinarize' per creare un'immagine binaria dall'immagine grezza da aspetti di controllo della soglia adattativa al valore di sensibilità di 0,59. Il fattore di sensibilità della soglia predefinito, 0,5, era basso e non era in grado di rilevare le macchie e le macchie più luminose dall'immagine, quindi l'abbiamo aumentato a 0,59.
L'immagine binarizzata viene quindi elaborata attraverso un filtro mediano utilizzando la funzione "medfilt2" poiché l'immagine binarizzata è bidimensionale. Impostiamo ogni pixel di output per contenere il valore mediano nell'intorno 5 x 5 attorno al pixel corrispondente nell'immagine binarizzata di input. Ciò riduce il rumore e conserva i bordi in un quadrato 5 x 5 attorno a ciascun pixel. Successivamente, applichiamo una finestra scorrevole usando 'strel', per creare un elemento strutturante piatto a forma di disco con un raggio di quartiere di 2 per identificare ogni pixel centrale, di origine, in ogni intorno di disco. Abbiamo utilizzato un elemento di struttura del disco perché stiamo analizzando ogni punto circolare e i pixel all'interno di ogni punto, quindi un elemento a forma di disco è più utile.
Una volta che l'immagine è stata filtrata, può essere pulita utilizzando la funzione "imclose" per rimuovere i punti neri tra i pixel bianchi filtrati nell'immagine e chiudere tutti gli spazi attorno ad essa. L'immagine completamente elaborata può quindi essere tracciata nella seconda sottotrama della figura preassegnata, consentendo un confronto tra l'immagine grezza e quella filtrata.
Passaggio 4: isolamento del tumore tramite maschera ellittica


I punti luminosi del tumore possono quindi essere isolati dall'immagine filtrata principale attraverso una maschera ellittica pre-generata. Per creare questa maschera, è necessario conoscere la dimensione dell'immagine di scansione MRI originale e grezza e, utilizzando la lunghezza della riga e della colonna, rispettivamente come coordinate x e y, assegnare le coordinate centrali per l'ellittica. Impostiamo l'asse y come asse maggiore con un raggio di 50 unità dal centro e l'asse minore con un raggio di 40 unità dal centro.
Abbiamo utilizzato la funzione MATLAB 'meshgrid' per generare un piano cartesiano con coordinate di griglia bidimensionali basate sulle coordinate contenute nei vettori da 1 alla lunghezza dell'asse xe da 1 alla lunghezza dell'asse y dell'immagine. Col è una matrice in cui ogni riga è una copia dell'asse x e Row è una matrice in cui ogni colonna è una copia dell'asse y. La griglia cartesiana rappresentata dalle coordinate Col e Row ha righe length(1:Y_Size) e colonne length(1:X_Size). Utilizzare gli indici di Col e Row generati dalla griglia cartesiana per determinare l'equazione dell'ellisse in base al raggio predeterminato e alle coordinate del centro. Il contorno ellittico può ora essere riempito con i pixel bianchi trovati dalle macchie tumorali.
Utilizzando la maschera ellittica pre-generata possiamo ritagliare il tumore specifico che si desidera analizzare dall'immagine filtrata. La maschera ellittica rileva quali punti si adattano logicamente al contorno dell'ellisse e accetta questo come un punto sull'immagine filtrata per essere accettabile come tumore. La funzione "bwareafilt" filtra quindi dall'immagine tutti gli altri oggetti al di fuori di questo tumore rilevato. Abbiamo utilizzato una finestra specifica di 500 per 4000 basata empiricamente sulle dimensioni di tutte le immagini. Abbiamo quindi applicato un'altra finestra scorrevole con "strel" come elemento strutturante a forma di disco piatto con un raggio di prossimità maggiore di 6, per chiudere gli spazi tra ciascun pixel bianco centrale all'interno del tumore rilevato. Il punto tumorale rilevato viene ulteriormente ripulito utilizzando "imclose" per eliminare ulteriormente i pixel neri e riempire tutti i fori con "imfill". Questo tumore elaborato può quindi essere visualizzato nella terza sottotrama nella trama preallocata per fornire un confronto tra il tumore isolato e le immagini originali e filtrate della scansione MRI.
Passaggio 5: delineazione del tumore
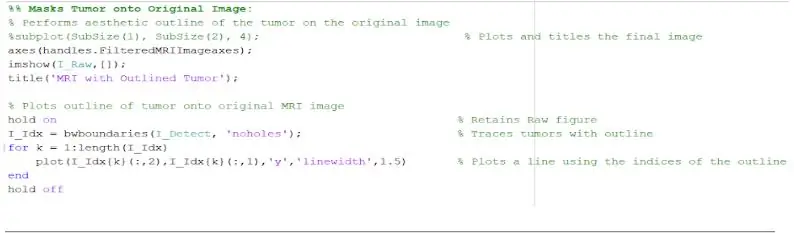
Ora che il tumore è isolato con la maschera, può essere delineato e visualizzato sull'immagine originale, per mostrarne la posizione esatta. Per fare ciò, abbiamo utilizzato la funzione "bwboundaries" per tracciare il tumore precedentemente rilevato con un contorno. Abbiamo specificato il contorno per non includere i fori all'interno dell'oggetto tumore mentre viene delineato. Questo può essere tracciato sull'immagine grezza originale, utilizzando un ciclo "for" che traccia il contorno attorno al tumore utilizzando gli indici della linea con una larghezza della linea di 1,5 pixel. Questo contorno viene quindi tracciato sull'immagine grezza, mostrando le dimensioni esatte e la posizione del tumore, rispetto alla scansione MRI originale.
Passaggio 6: analisi delle proprietà fisiche dei tumori
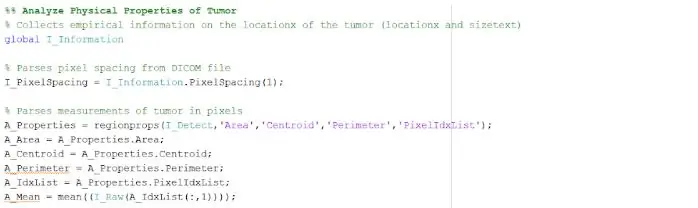
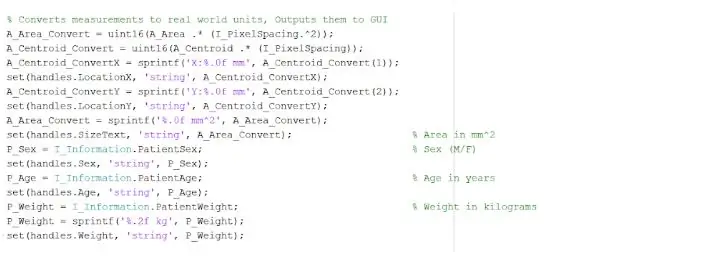
Il punto isolato e delineato può fornirci informazioni utili sulla dimensione, l'area e la posizione del tumore. Abbiamo utilizzato la funzione "regionprops" per rilevare le proprietà del tumore relative all'area, al perimetro, ai centroidi e al valore dell'indice di pixel. Questo valore dell'indice di pixel ci fornisce le unità del mondo reale per ogni pixel di ogni immagine, univoco per ogni scansione. Queste proprietà possono quindi essere convertite in unità reali di millimetri. Le informazioni empiriche che il programma ci fornisce sono uniche per ogni scansione MRI ed è estremamente utile per determinare la dimensione, la posizione e il tipo di tumore, che gli utenti possono analizzare e incorporare nell'interfaccia utente grafica.
Consigliato:
Rilevamento del movimento utilizzando MPU-6000 e Particle Photon: 4 passaggi

Rilevamento del movimento tramite MPU-6000 e fotone di particelle: MPU-6000 è un sensore di rilevamento del movimento a 6 assi con accelerometro a 3 assi e giroscopio a 3 assi incorporati. Questo sensore è in grado di tracciare in modo efficiente la posizione esatta e la posizione di un oggetto nel piano tridimensionale. Può essere impiegato i
Rilevamento del movimento utilizzando MPU-6000 e Raspberry Pi: 4 passaggi

Rilevamento del movimento utilizzando MPU-6000 e Raspberry Pi: MPU-6000 è un sensore di rilevamento del movimento a 6 assi con accelerometro a 3 assi e giroscopio a 3 assi incorporati. Questo sensore è in grado di tracciare in modo efficiente la posizione esatta e la posizione di un oggetto nel piano tridimensionale. Può essere impiegato i
Telecamera IP con rilevamento del volto utilizzando la scheda ESP32-CAM: 5 passaggi

Telecamera IP con rilevamento del volto utilizzando la scheda ESP32-CAM: questo post è diverso dagli altri e diamo un'occhiata alla scheda ESP32-CAM molto interessante che è sorprendentemente economica (meno di $ 9) e facile da usare. Creiamo una semplice telecamera IP che può essere utilizzata per lo streaming di un feed video in diretta utilizzando il 2
Robot di Halloween con rilevamento del volto e rilevamento del sorriso: 8 passaggi (con immagini)

Monitoraggio del viso e rilevamento del sorriso Robot di Halloween: Halloween sta arrivando! Abbiamo deciso di costruire qualcosa di interessante. Incontra i robot Ghosty e Skully. Possono seguire il tuo viso e sanno quando sorridi per ridere con te! Questo progetto è un altro esempio dell'utilizzo dell'app iRobbie che converte l'iPhone in
Realizza un dispositivo di rilevamento e rilevamento a ultrasuoni a casa: 7 passaggi (con immagini)

Crea un dispositivo di rilevamento e distanza ad ultrasuoni a casa: ciao! Sono Sourabh Kumar, ero ansioso di creare un radar allarmante ma non ci proverò di nuovo, ma oggi ti guiderò a fare un rilevamento a ultrasuoni e un dispositivo di rilevamento a casa usando un sensore a ultrasuoni (ricetrasmettitore) so che ce ne sono molti professionista
