Sommario:
- Passaggio 1: organizzazione dei dati della mammografia
- Passaggio 2: elaborazione delle immagini
- Passaggio 3: codice di soglia
- Passaggio 4: ricerca di anomalie per ogni immagine binaria
- Passaggio 5: tracciare la posizione e le dimensioni della massa diagnosticata per il confronto visivo
- Passaggio 6: implementazione del secondo metodo di confronto
- Passaggio 7: analisi dei dati raccolti
- Passaggio 8: creare il proprio classificatore
- Passaggio 9: miglioramenti? Qualche idea?
- Autore John Day day@howwhatproduce.com.
- Public 2024-01-30 10:02.
- Ultima modifica 2025-01-23 14:49.
Lo scopo di questo progetto era identificare e utilizzare un parametro per elaborare le immagini mammografiche in scala di grigi di varie classificazioni di tessuti di fondo: tessuto grasso, ghiandolare grasso e tessuto denso. Questa classificazione viene utilizzata quando i radiologi analizzano le mammografie e devono considerare se la densità dei tessuti oscurerà eventuali anomalie come lesioni o tumori. Questo perché entrambe le normali strutture fisiologiche come il tessuto ghiandolare e il tessuto connettivo fibroso. e morfologie anomale come calcificazioni e tumori appariranno molto luminose sulla mammografia mentre il tessuto adiposo meno denso apparirà nero. Pertanto, era opportuno programmare un classificatore in grado di manipolare i livelli di intensità dei pixel per visualizzare e identificare al meglio le masse.
Passaggio 1: organizzazione dei dati della mammografia
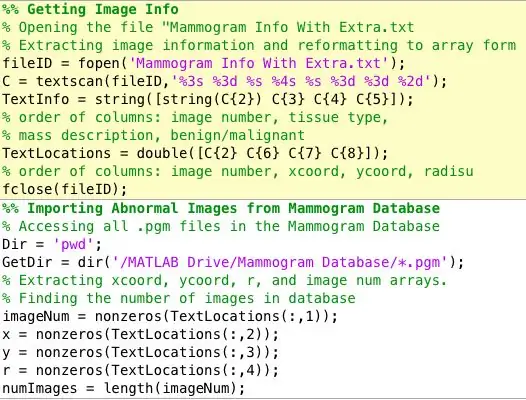
Una delle prime cose che ho capito di dover gestire è stata organizzare i dati in modo molto chiaro, conciso e accessibile. Queste sono le variabili che ho estratto dal database mini-MIAS delle mammografie. Ho creato due array. Uno contenente 4 colonne:
- Numero immagine:
- coordinata x di massa
- y coordinata di massa
- Raggio di massa: (Questo ha definito una dimensione approssimativa per la massa
Il secondo array conteneva informazioni sulla classificazione:
- Tipo di tessuto di fondo: grasso (F), ghiandolare grasso (G), denso (D)
- Descrizione della massa: ben definita (CIRC), spiculata (SPIC), altro mal definita (MISC) distorsione architettonica (ARCH), asimmetria (ASYM), normale (NORM)
- Diagnosi: benigna (B), maligna (M)
Poiché lo scopo di questo progetto era determinare la soglia migliore per ciascun tipo di tessuto di fondo, non tutte le informazioni erano necessarie. Tuttavia, puoi espandere il tuo progetto per includere l'analisi della trama e testare il tuo classificatore rispetto alle descrizioni di massa note.
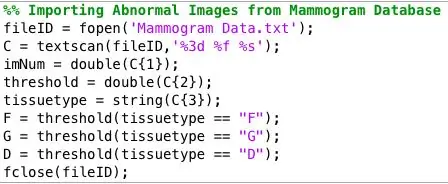
Nota a margine: il database da cui ho ottenuto le immagini mammografiche diagnosticate ha organizzato le informazioni su ciascuna mammografia in un file di testo separato dalle immagini. È stato leggermente difficile per me estrarre i dati da un file di testo e organizzarli in moduli di array, ma il seguente collegamento è stato molto utile per capire tutto questo. In alternativa, regola semplicemente il codice che ho incollato sopra per i tuoi scopi.
Formato file mammografia: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
TextScan Help: https://www.mathworks.com/help/matlab/ref/textsca…Database mammogrammi:
Passaggio 2: elaborazione delle immagini
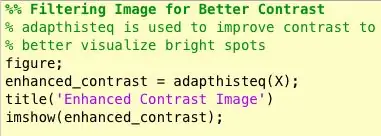
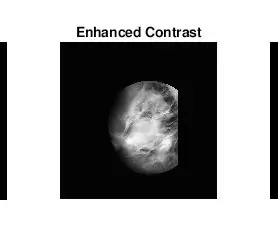
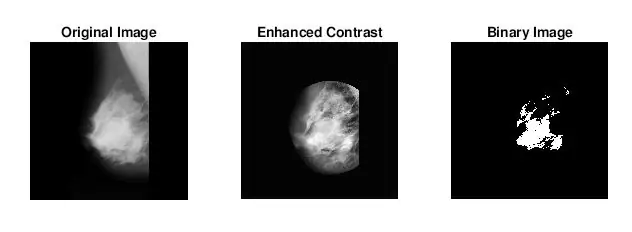
Bene, la seconda cosa che è venuta fuori quando stavo cercando di identificare le masse era che per molte mammografie anormali non potevo dire visivamente dove fosse l'anomalia o quanto fosse grande. Ovviamente, non essendo un radiologo esperto, era previsto. Tuttavia, il modo più semplice per trovare le anomalie (secondo le mie lunghe ricerche su Google) era guardare le concentrazioni di aree luminose e scure. Ho utilizzato principalmente la funzione Adapthisteq per migliorare il contrasto dell'immagine e quindi imbinarizzare per convertire l'immagine in un'immagine binaria per sperimentare diversi livelli di soglia.
- Adapthisteq: questa funzione trasforma i valori di intensità delle immagini in scala di grigi e RGB utilizzando l'equalizzazione dell'istogramma adattivo a contrasto limitato. In altre parole, adatta l'istogramma dei valori di intensità a un tipo di distribuzione specificato. Il collegamento mathworks per questa funzione è allegato di seguito per ulteriori letture.
- imbinarize: crea un'immagine binaria da un'immagine in scala di grigi assegnando tutti i pixel sopra una certa intensità a 1s e i pixel sotto quel valore a 0. Ho usato questa funzione per testare la soglia ottimale per ridurre il rumore di fondo del tessuto.
Passaggio 3: codice di soglia
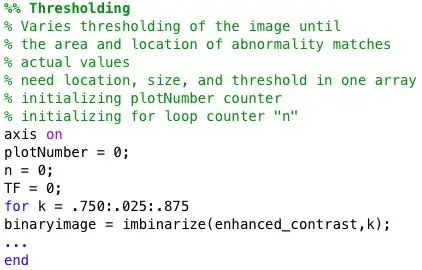
Un ciclo for viene utilizzato per binarizzare la mammografia con soglie variabili. Per dare una visione più ampia dell'immagine, il ciclo for contiene il codice dal passaggio 3 al passaggio 7. Quindi ogni immagine binaria verrà analizzata per anomalie. Inoltre, questo ciclo for è racchiuso in un altro ciclo for che importa una nuova immagine mammografica dal database in ogni iterazione.
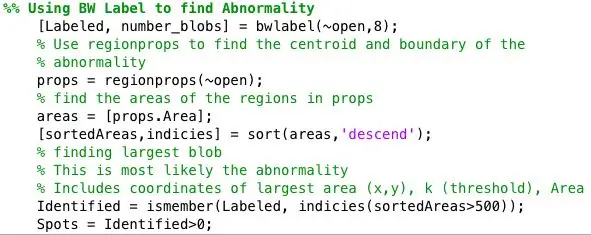
Passaggio 4: ricerca di anomalie per ogni immagine binaria
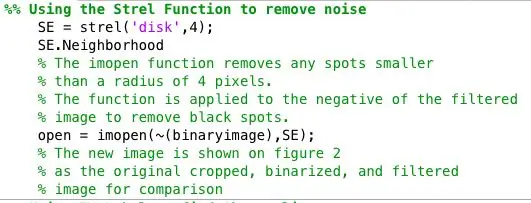
Ho ulteriormente elaborato le immagini binarie utilizzando la funzione strel insieme a imopen per rimuovere il rumore di fondo. L'immagine binaria del passaggio precedente viene invertita e filtrata utilizzando l'intorno definito da SE. Quindi ho usato bwlabel per etichettare tutte le aree che avevano almeno 8 pixel collegati.
La funzione region props è stata utilizzata per trovare il centroide e le proprietà dell'area di ogni punto identificato da bwlabel.
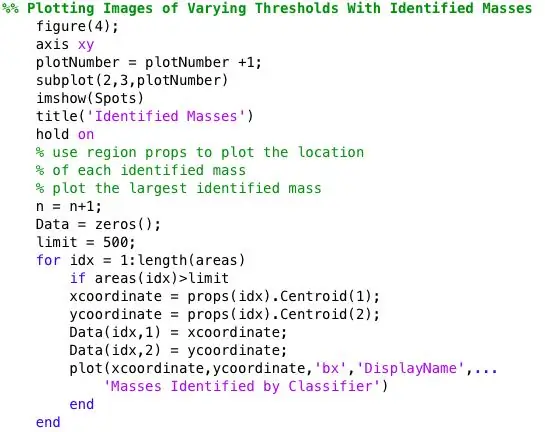
Quindi tutti gli spot più grandi di 500 pixel sono stati identificati utilizzando ismember. I centroidi per i punti identificati sono stati tracciati su un'immagine che mostrava solo i punti più grandi nell'area di 500. Area Identified = ismember(Labeled, indicies(sortedAreas>500)); Spot = Identificato>0;
Passaggio 5: tracciare la posizione e le dimensioni della massa diagnosticata per il confronto visivo
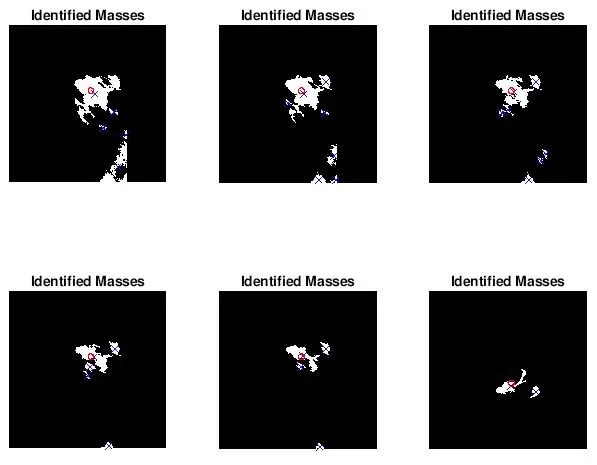

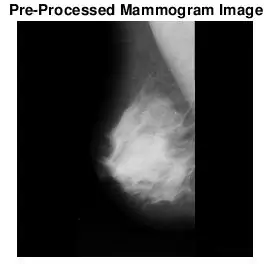
Volevo vedere se le macchie trovate da bwlabel erano corrette. L'ho fatto in due modi. Per prima cosa ho analizzato l'accuratezza del mio classificatore facendo un confronto visivo. Ho semplicemente tracciato la dimensione effettiva e la posizione dell'anomalia (cerchio rosso) e la posizione determinata dal codice (x blu) sull'immagine mammografica pre-elaborata. Le sei immagini sopra mostrano gli effetti dell'aumento del valore di soglia della scala di grigi.
Passaggio 6: implementazione del secondo metodo di confronto

Il secondo modo in cui ho testato il classificatore e i valori di soglia consisteva nel determinare se le posizioni trovate dal classificatore si trovavano entro una certa distanza dalle coordinate dell'anomalia diagnosticata. Ho salvato le soglie per le quali almeno uno dei punti identificati era entro 1,5*r dall'anomalia nota in un file di testo separato chiamato Mammogram Data. Lo scopo era trovare la soglia minima necessaria per il mio classificatore per identificare l'anomalia.
Passaggio 7: analisi dei dati raccolti
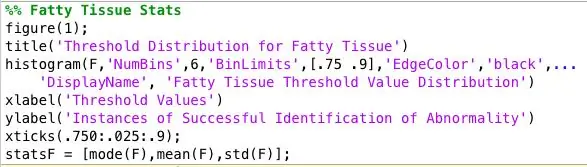
Ho eseguito il programma su tutte le immagini anormali della mammografia e mi è rimasto un enorme file di testo di dati. Per trovare la soglia migliore per ogni tipo di tessuto ho organizzato i dati per tipo di tessuto e tracciato un istogramma dei valori di soglia per ogni tipo di tessuto. Il valore di soglia corretto è stato deciso su quale soglia ha fornito i risultati più accurati per ciascun tipo di tessuto. Ho salvato questi dati per caricarli nel mio classificatore.
Passaggio 8: creare il proprio classificatore

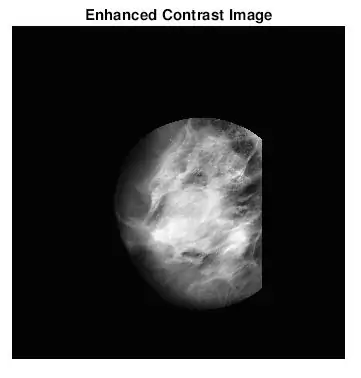
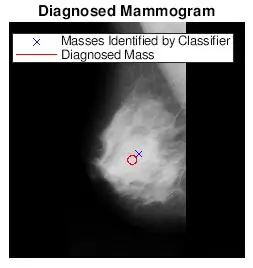
Dopo aver trovato i valori di soglia più appropriati per ciascun tipo di tessuto, ho modificato il mio codice originale in modo che un utente inserisse il numero dell'immagine e il tipo di tessuto per scegliere la soglia per l'immagine della mammografia. Ho quindi tracciato la posizione della mammografia diagnosticata con le posizioni trovate sulle immagini della mammografia originale. Volevo renderlo più divertente, quindi ho programmato una funzione per ritagliare una regione circolare che circonda il ROI. L'utente verrà istruito a scegliere un punto centrale e diversi punti che meglio racchiudono il ROI. Ho allegato entrambi i file matlab qui.
Passaggio 9: miglioramenti? Qualche idea?
Mentre stavo scrivendo questa guida, ho iniziato a vedere molti miglioramenti che potevo apportare al classificatore, come trovare modi per distinguere tra diversi tipi di masse identificate in base all'analisi della trama o migliorare la mia sezione di test per la precisione del SandBoxProject. file. Dato che questo era un progetto con una scadenza, ho dovuto fermarmi da qualche parte, ma spero di essere in grado di utilizzare le capacità di elaborazione delle immagini che ho appreso in altre applicazioni. Inoltre, ho allegato il file che è stato utilizzato per elaborare in batch tutte le immagini mammografiche anomale.
Consigliato:
Come effettuare la registrazione della temperatura e dell'intensità della luce - Simulazione Proteus - Fritzing - Liono Maker: 5 passaggi

Come effettuare la registrazione della temperatura e dell'intensità della luce | Simulazione Proteus | Fritzing | Liono Maker: Ciao, sono Liono Maker, questo è il mio canale YouTube ufficiale. Questo è un canale YouTube open source. Ecco il link: Canale YouTube di Liono Maker ecco il link del video: Temp & registrazione dell'intensità della luceIn questo tutorial impareremo come creare Temper
Ottica nelle sculture di luce: 8 passaggi (con immagini)

L'ottica nelle sculture di luce: Ciao, mi chiamo Julien Hogert. Mi sono diplomato qualche anno fa alla scuola di cinema “Louis Lumière”, dove ho studiato, tra l'altro, fotografia e molta ottica. Ora lavoro nel cinema, ma faccio anche sculture con le luci. Negli ultimi 3 anni
Macchina da disegno Polargraph su larga scala con testa della penna retrattile: 4 passaggi (con immagini)
Macchina da disegno Polargraph su larga scala con penna retrattile: *L'installazione su larga scala di questa macchina è stata concepita ed eseguita con Rui Periera Questo è un progetto per il disegno open source Polargraph (http://www.polargraph.co.uk/) progetto. È dotato di una testa della penna retrattile e hardware per consentirgli di
Monitor LCD per l'utilizzo della CPU e della RAM Arduino: 5 passaggi

Monitor LCD per l'utilizzo della CPU + RAM Arduino: Ciao a tutti, oggi ho realizzato un monitor per l'utilizzo della CPU + RAM Arduino utilizzando un semplice schizzo Arduino e un programma VB.net. In questo tutorial ti mostrerò come realizzarlo. Il programma vb.net dispone di un tester di connessione Arduino e puoi scrivere testo personalizzato su
N: Come realizzare una scultura a più strati in acrilico e LED con livelli di illuminazione variabili: 11 passaggi (con immagini)

N: Come realizzare una scultura multistrato in acrilico e LED con livelli di illuminazione variabili: qui puoi scoprire come personalizzarti come realizzato per la mostra www.laplandscape.co.uk curata dal gruppo di arte/design Lapland. Altre immagini possono essere viste su flickrQuesta mostra va da mercoledì 26 novembre a venerdì 12 dicembre 2008 incluso
