Sommario:
- Autore John Day day@howwhatproduce.com.
- Public 2024-01-30 10:00.
- Ultima modifica 2025-01-23 14:49.
Di: Phuc Lam, Paul Yeung, Eric Reyes
Riconoscere che gli errori nella segmentazione dei polmoni produrranno false informazioni sull'identificazione di un'area di malattia e possono influenzare direttamente il processo di diagnosi. Le moderne tecniche di ausilio al computer non sono riuscite a fornire risultati accurati quando le malattie polmonari hanno forme difficili. Queste forme anormali possono essere causate da versamenti pleurici, consolidamenti, ecc. Applicando la tecnica di segmentazione polmonare, in cui i confini del polmone sono isolati dal tessuto toracico circostante, la nostra app può identificare i confini con le soglie di input dell'utente per fornire visualizzazioni completamente personalizzabili delle forme dei polmoni, Lo scopo di questo progetto MatLab è creare un'app di segmentazione polmonare interattiva di facile utilizzo per rilevare le condizioni patologiche delle immagini a raggi X dei polmoni. Il nostro obiettivo è creare un modo più efficace per illustrare e identificare i polmoni anormali al fine di offrire a medici e radiologi un modo più affidabile per diagnosticare le malattie polmonari. Utilizzando lo strumento di progettazione app in MatLab, il programma è progettato per funzionare specificamente con le scansioni radiografiche del torace e tomografia computerizzata (TC), ma è anche testato per funzionare con le scansioni MRI.
Le istruzioni seguenti contengono la nostra tecnica di filtraggio del rumore (filtro di Wiener passa basso) e la soglia dell'immagine (utilizzando l'istogramma di intensità dell'immagine in scala di grigi) e utilizzando un gradiente morfologico (la differenza tra la dilatazione e l'erosione di un'immagine) per identificare una regione di interesse. L'istruzione spiegherà quindi come integriamo tutti gli elementi nell'interfaccia utente grafica (GUI).
Nota:
1). Questo progetto è ispirato da un documento di ricerca: "Segmentazione e analisi dell'immagine dei polmoni anormali alla TC: approcci attuali, sfide e tendenze future". Che puoi trovare qui
2). Stiamo usando immagini a raggi X da NIH: Clinical Center. Il link lo trovate qui
3). L'aiuto per la progettazione dell'app può essere trovato qui
4). Prima di eseguire il codice: è necessario modificare il percorso Dir (nella riga 34) nella directory dei file e il tipo di immagine (riga 35) (stiamo analizzando *.png).
Passaggio 1: Passaggio 1: caricamento dell'immagine
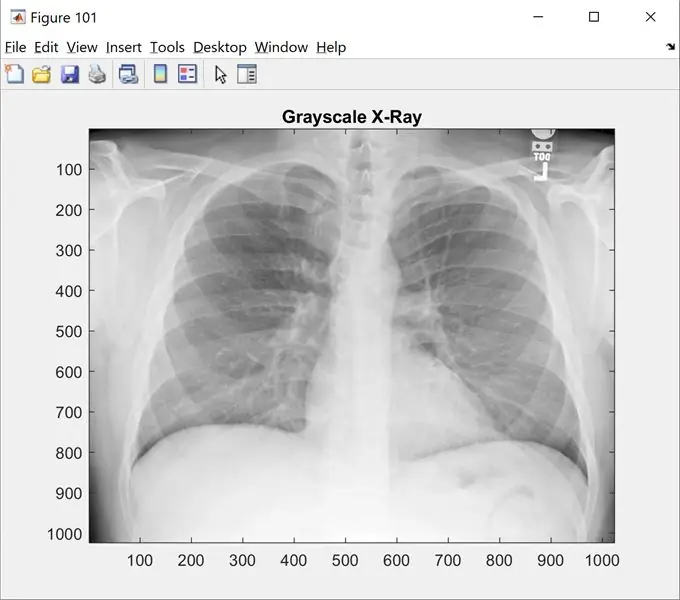
Questo passaggio ti mostrerà l'immagine originale in scala di grigi. Cambia il 'nome_di_immagine.png' con il nome della tua immagine
chiaro; clc; chiudi tutto;
%% Caricamento immagini
raw_x_ray='nome_di_immagine.png';
I=imread(raw_x_ray);
figura(101);
imshow(I);
mappa colore(grigio);
title('Radi X in scala di grigi');
Passaggio 2: Passaggio 2: filtraggio del rumore e istogramma
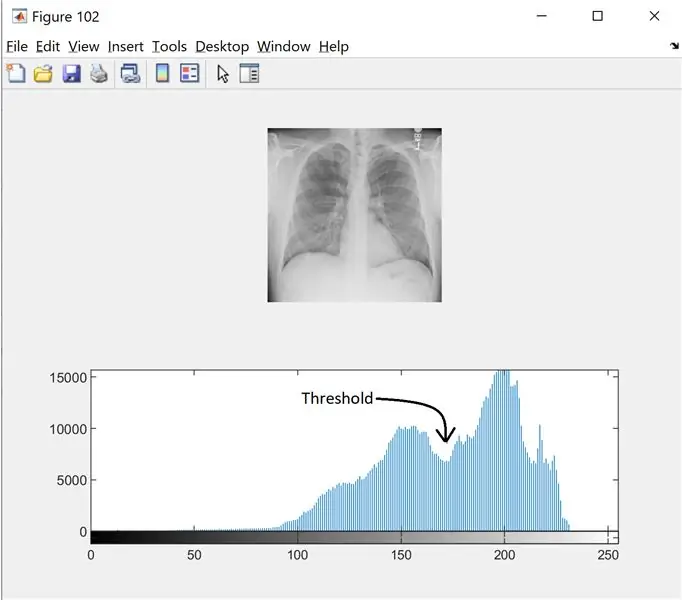
Per trovare la soglia per l'immagine in scala di grigi, osserviamo l'istogramma per vedere se le modalità sono distinte. Leggi di più qui
I=wiener2(I, [5 5]);
figura(102);
sottotrama (2, 1, 1);
imshow(I);
sottotrama (2, 1, 2);
imhist(I, 256);
Passaggio 3: Passaggio 3: impostazione delle soglie
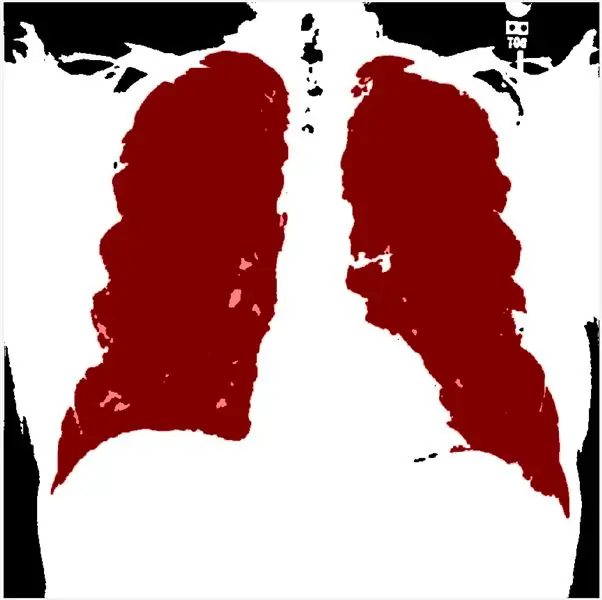
Questo passaggio consente di impostare la soglia in base all'istogramma. morphologicalGradient evidenzierà la regione di interesse in rosso e la funzione visboundaries sovrapporrà l'immagine delineata e filtrata del polmone in rosso.
Usando regionprops, possiamo esigere gli array di solidità e ordinarli in ordine decrescente. Quindi binario l'immagine di gray sclae e applico il metodo del gradiente morfologico e mLoren Shurasking per evidenziare la regione di interesse (ROI). Il prossimo passo è invertire l'immagine in modo che la ROI del polmone sia bianca su sfondo nero. Uso la funzione showMaskAsOverlay per visualizzare 2 mask. Nota: il codice è ispirato a Loren Shure, link.
Infine, creo un contorno rosso utilizzando bwbwboundaries e mascherando l'immagine del filtro e i bordi.
a_thresh = io >= 172; % imposta questa soglia
[labelImage, numberOfBlobs] = bwlabel(a_thresh);
props = regionprops(a_thresh, 'all');
sortedSolidity = sort([props. Solidity], 'descend');
SB = sortedSolidity(1);
se SB == 1 % SB accetta solo solidità == 1 filtra le ossa
binaryImage = imbinarize(I); figura(103);
imshow(binaryImage); mappa colore(grigio);
SE = strel('quadrato', 3);
morphologicalGradient = imsubtract(imdilate(binaryImage, SE), imerode(binaryImage, SE));
mask = imbinarize(Gradiente morfologico, 0,03);
SE = strel('quadrato', 2);
maschera = imclose(maschera, SE);
mask = imfill(maschera, 'buchi');
maschera = bwareafilt(maschera, 2); % controllo numero di area mostra
notMask = ~maschera;
maschera = maschera | bwpropfilt(notMask, 'Area', [-Inf, 5000 - eps(5000)]);
showMaskAsOverlay(0.5, mask, 'r'); % devi scaricare app/funzione showMaskAsOverlay
BW2 = imfill(binaryImage, 'buchi');
nuova_immagine = BW2;
nuova_immagine(~maschera) = 0; % inverti sfondo e buchi
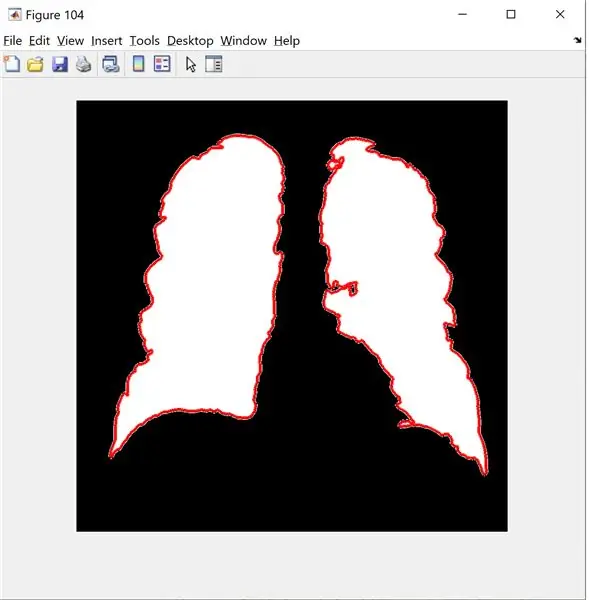
B=bwboundaries(new_image); % può accettare solo 2 dimensioni
figura(104);
imshow(new_image);
aspettare
confini visivi (B);
fine
Passaggio 4: creazione della GUI
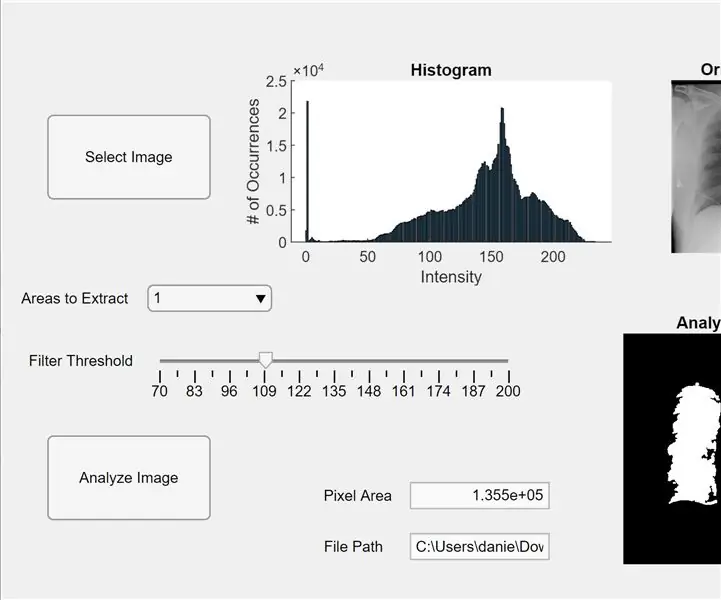
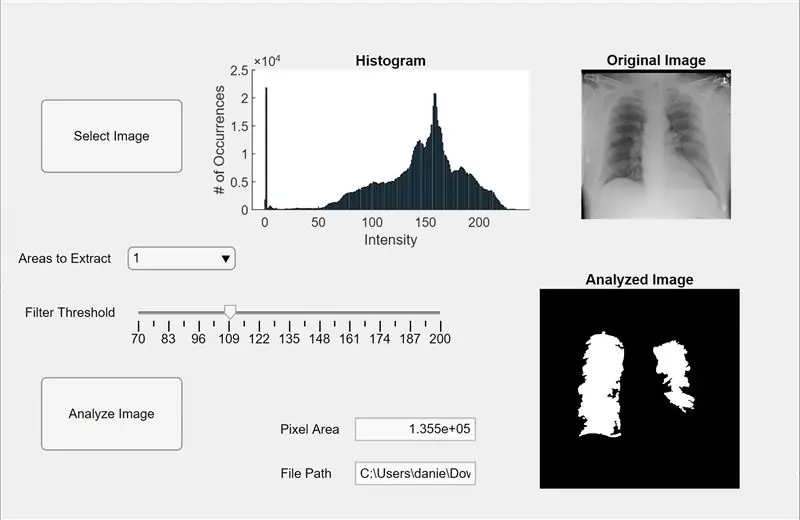
Ora integriamo il codice precedente in un'app MATLAB. Apri App Designer in MATLAB (Nuovo > App). Innanzitutto, progettiamo l'interfaccia facendo clic e trascinando su tre assi nell'area di lavoro centrale. Successivamente, facciamo clic e trasciniamo due pulsanti, un campo di modifica (testo), un campo di modifica (numerico), un dispositivo di scorrimento e un menu a discesa. Ciascuno di due assi visualizzerà l'anteprima e analizzerà l'immagine, mentre il terzo asse visualizzerà un istogramma di pixel per l'anteprima dell'immagine “selezionata”. La casella del campo di modifica (testo) visualizzerà il percorso del file dell'immagine selezionata e il campo di modifica (numerico) visualizzerà l'area dei pixel rilevati dei polmoni.
Ora passa dalla visualizzazione struttura alla visualizzazione codice in App Designer. Inserisci nel codice il codice per le proprietà facendo clic sul pulsante rosso "Proprietà" con un segno più. Inizializzare le proprietà I, soglia e regionToExtract come nel codice fornito di seguito. Quindi, fai clic con il pulsante destro del mouse su un pulsante nella parte in alto a destra dell'area di lavoro (il Browser dei componenti) e vai da Richiamate> Vai a… richiamata. Aggiungi il codice per "funzione SelectImageButtonPushed(app, event)." Questo codice ti permette di selezionare un'immagine da analizzare dal tuo computer usando uigetfile. Dopo aver selezionato un'immagine, apparirà un'immagine di anteprima sotto gli assi accompagnata da un istogramma. Quindi, fai clic con il pulsante destro del mouse sull'altro pulsante e ripeti la stessa procedura per creare una funzione di callback.
Aggiungi il codice sotto "function AnalyzeImageButtonPushed(app, event)." Questo codice eseguirà il conteggio dei pixel e il rilevamento dei blob sull'immagine di anteprima sul pulsante di analisi dell'immagine (quello su cui hai fatto clic con il pulsante destro del mouse per questo codice). Dopo aver programmato i pulsanti, andremo ora a programmare lo slider e il menu a tendina. Fare clic con il pulsante destro del mouse sul dispositivo di scorrimento, creare una funzione di callback e aggiungere il codice sotto "function FilterThresholdSliderValueChanged(app, event)" fino alla fine. Ciò consente al dispositivo di scorrimento di regolare la soglia dell'intensità del grigio.
Crea una funzione di callback per il menu a discesa e aggiungi il codice sotto "function AreastoExtractDropDownValueChanged(app, event)" per consentire al menu a discesa di modificare il numero di blob visualizzati sugli assi dell'immagine analizzata. Ora, fai clic su ciascuna entità nel Browser dei componenti e modifica le loro proprietà a tuo piacimento, come cambiare i nomi delle entità, rimuovere gli assi e modificare la scala. Trascina e rilascia le entità del Browser dei componenti in Visualizzazione struttura in un layout funzionale e di facile comprensione. Ora hai un'app in MATLAB che può analizzare le immagini dei polmoni per l'area dei pixel!
proprietà (Accesso = privato)I = ; % file immagine
soglia = 257; %soglia per binarizzare l'intensità del grigio
regionToExtract = 2;
fine
funzione SelectImageButtonPushed (app, evento)
clc;Dir = 'C:\Users\danie\Downloads\images_004\images'; %define file invariato "prefisso"
[imageExt, percorso] = uigetfile('*.png'); %prendi la parte variabile del nome dell'immagine
imageName = [Dir fileep imageExt]; %concatena stringhe invariate e variabili
app. I = imread(nomeimmagine); %leggi l'immagine
imshow(app. I, 'genitore', app. UIAxes); %visualizza l'immagine
app. FilePathEditField. Value = percorso; %visualizza il percorso del file da cui proviene l'immagine originale
fine
funzione AnalyzeImageButtonPushed(app, event)
originalImage = app. I;
originalImage = wiener2(app. I, [5 5]); % filtro per la rimozione dei punti
histogram(app. AxesHistogram, app. I, 256); %visualizza l'istogramma dell'immagine
a_thresh = originalImage >= app.threshold; % imposta questa soglia
labelImage = bwlabel(a_thresh);
props = regionprops(a_thresh, 'all');
sortedSolidity = sort([props. Solidity], 'descend');
SB = sortedSolidity(1);
se SB == 1 % SB accetta solo solidità ==1 filtra le ossa
SE = strel('quadrato', 3);
morphologicalGradient = imsubtract(imdilate(labelImage, SE), imerode(labelImage, SE));
mask = imbinarize(Gradiente morfologico, 0,03);
SE = strel('quadrato', 2);
maschera = imclose(maschera, SE);
mask = imfill(maschera, 'buchi');
mask = bwareafilt(mask, app.regionsToExtract);
% controllo numero di area mostra
notMask = ~maschera;
maschera = maschera | bwpropfilt(notMask, 'Area', [-Inf, 5000 - eps(5000)]);
BW2 = imfill(labelImage, 'holes');
nuova_immagine = BW2;
nuova_immagine(~maschera) = 0;
B = bwboundaries(new_image); % può accettare solo 2 dimensioni imshow(new_image, 'parent', app. UIAxes2);
hold(app. UIAxes2, 'on');
confini visivi (B);
set(gca, 'YDir', 'reverse');
lungArea = bwarea(new_image);
app. PixelAreaEditField. Value = lungArea;
fine
fine
function FilterThresholdSliderValueChanged(app, event)
app.threshold = app. FilterThresholdSlider. Value;
fine
function AreastoExtractDropDownValueChanged(app, event)stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double(stringNumber);
fine
fine
Consigliato:
MATLAB Easy Face Detection: 4 passaggi

MATLAB Easy Face Detection: l'obiettivo principale di queste istruzioni è mostrare quanto facile sarà l'elaborazione delle immagini, con l'aiuto di MATLABFace detection e tracking è stato un campo di ricerca importante e attivo, ecco perché spiegherò come si può fare con
Come codificare e pubblicare Matlab 2016b in Word (guida per principianti): 4 passaggi

Come codificare e pubblicare Matlab 2016b in Word (guida per principianti): Matlab è un programma di linguaggio ad alte prestazioni utilizzato per calcolare risultati tecnici. Ha la capacità di integrare immagini, calcoli e programmazione in modo intuitivo. Con questo programma, l'utente può pubblicare problemi e soluzioni
Controllare Raspberry Pi usando Matlab: 5 passaggi

Controllo di Raspberry Pi usando Matlab: Ehi, questo tutorial riguarda il controllo del tuo Raspberry Pi senza testa usando Matlab. Potrebbe essere necessario installare matlab alla versione più recente per il supporto alla nuova scheda raspberry pi
Controller robotico ROS basato su Matlab: 9 passaggi
Controller robotico ROS basato su Matlab: sin da quando ero bambino, ho sempre sognato di essere Iron Man e continuo a farlo. Iron Man è uno di quei personaggi che è realisticamente possibile e in poche parole aspiro a diventare Iron Man un giorno anche se la gente ride di me o dice che è impossibile
Miglioramenti alla programmazione del treno in MATLAB: 6 passaggi

Miglioramenti alla programmazione dei treni in MATLAB: Obiettivo: l'obiettivo di questo sistema programmato è guardare un Arduino su piccola scala e applicare la codifica su una scala più grande per migliorare potenzialmente alcune caratteristiche di sicurezza dei sistemi Amtrak Railroad. Per fare questo, abbiamo aggiunto un sensore di umidità del suolo
